еѓЉиѓї
еЕНзЦЂзїДжµЛеЇПпЉМзЫЄиЊГдЇОеЕ®еЯЇеЫ†зїДжµЛеЇПгАБеЕ®е§ЦжШЊе≠РжµЛеЇПгАБеЯЇеЫ†panelжЭ•иѓіпЉМе§ІеЃґдЉЪжШЊеЊЧйЩМзФЯеТМжѓФиЊГйЪЊзРЖиІ£гАВзЃАеНХи®АдєЛпЉМеЕНзЦЂзїДеЕґеЃЮжШѓжЯРзІНз®ЛеЇ¶зЪДжНХиОЈеЯЇеЫ†зїДжµЛеЇПпЉМдєЯжШѓдЄАзІНеЯЇеЫ†panelпЉМеЕ≥ж≥®зЪДжШѓ 608-665 IG and TR genesпЉМињЩжЃµеЯЇеЫ†еЕЈжЬЙиЊГйЂШе§Ъж†ЈжАІпЉМзРЖиЃЇдЄКиГљиЊЊ1016~1018зЪДзІНз±ї гАВеМЇеИЂеЬ®дЇОжЩЃйАЪзЪДеЯЇеЫ†panelйЬАи¶БеЬ®жѓПдЄ™еЯЇеЫ†зЪДжѓПдЄ™е§ЦжШЊе≠РдЄКйЭҐиЃЊиЃ°жОҐйТИпЉМдњЭиѓБеЕ®жЦєдљНзЪДи¶ЖзЫЦпЉМиАМеЕНзЦЂзїДйЬАи¶БиЃЊиЃ°жЬЙйЩРеЉХзЙ©и¶ЖзЫЦйЂШе§Ъж†ЈжАІзЪД¬†IG¬†еТМTRгАВ
еЕ≥дЇОеЕНзЦЂзїДTзїЖиГЮеПЧдљУпЉИTCRпЉЙеТМBзїЖиГЮеПЧдљУпЉИBCRпЉЙзЪДеЯЇз°АзЯ•иѓЖиѓ¶иІБеПѓеПВиАГTCR/BCRеЕНзЦЂзїДжµЛеЇПдЄОеЇФзФ®жЦєеРСгАВ
зђђдЄАйГ®еИЖпЉЪиЙЊж≤РиТљImmuHub¬Ѓ иЙЊж≤РиТљеЉАеПСзЪДImmuHub¬ЃжКАжЬѓеє≥еП∞еИ©зФ®5’RACEпЉИRNAйАВзФ®пЉЙжИЦе§ЪйЗНPCRжКАжЬѓпЉИDNAйАВзФ®пЉЙпЉМдљњжИСдїђиГљзЫіжО•дїОеЯЇеЫ†зїДRNAжИЦDNAжЭ•жЙ©еҐЮTCR/BCRзЪДеЕ®йХњеЇПеИЧжИЦCDR3еМЇеЇПеИЧпЉИжЦЗеЇУжЮДеїЇпЉЙпЉМдїОиАМињЫи°МйЂШйАЪйЗПжµЛеЇПеТМжХ∞жНЃеИЖжЮРгАВ еЫЊ1 жХ∞жНЃеИЖжЮРеИЖдЄЇеЯЇз°АеИЖжЮРдЄОжЈ±еЇ¶еИЖжЮРгАВеЬ®ж†ЗеЗЖеИЖжЮРжК•еСКдЄ≠пЉМиЙЊж≤РиТљжПРдЊЫдЇЖдЄЙзІНжЈ±еЇ¶еИЖжЮРпЉЪV-JеЯЇеЫ†зЙЗжЃµзїДеРИеЫЊпЉМTop10 CDR3еЇПеИЧйХњеЇ¶еИЖеЄГжЯ±зКґеЫЊеТМеЕЛйЪЖйҐСзОЗеИЖз±їеСИзО∞гАВ еѓєдЄЛжЬЇжµЛеЇПжХ∞жНЃињЫи°Миі®йЗПиѓДдЉ∞пЉМеОїйЩ§еОЯеІЛжХ∞жНЃпЉИraw dataпЉЙдЄ≠зЪДжЧ†жХИеЇПеИЧпЉМиОЈеЊЧжЬЙжХИжµЛеЇПжХ∞жНЃпЉИclean dataпЉЙгАВињЗжї§еРОжХ∞жНЃдЄОеЫљйЩЕеЕНзЦЂйБЧдЉ†е≠¶жХ∞жНЃеЇУпЉИIMGTпЉМhttp://www.imgt.orgпЉЙ*еЇПеИЧжѓФеѓєпЉМиОЈеЊЧжѓПжЭ°еЇПеИЧдЄ≠VгАБDгАБJеТМCеЯЇеЫ†зЙЗжЃµIDпЉМзДґеРОеЬ®еЕЈжЬЙеРИж†ЉзЪДCDR3еМЇеЯЯзЪДеЯЇз°АдЄКпЉМйЙіеЃЪеЗЇеЕЛйЪЖзЪДCDR3ж†ЄйЕЄеЇПеИЧдї•еПКж∞®еЯЇйЕЄеЇПеИЧпЉМеєґеѓєеЊЧеИ∞зЪДжѓПзІНеЕЛйЪЖињЫи°МиЃ°жХ∞еТМйҐСзОЗзЪДиЃ°зЃЧгАВзїУжЮЬз§ЇдЊЛе¶ВдЄЛеЫЊжЙАз§ЇпЉЪ пЉИ*IMGTжХ∞жНЃеЇУзФ®дЇОеИЖжЮРжКЧеОЯеПЧдљУгАБеЕНзЦЂзРГиЫЛзЩљпЉИжКЧдљУпЉЙзЪДеЯЇеЫ†пЉИз≠ЙдљНеЯЇеЫ†пЉЙдї•еПКTзїЖиГЮеПЧдљУпЉМжХізРЖдїОй±Љз±їеИ∞дЇЇз±їз≠ЙйҐМй™®иДКж§ОеК®зЙ©зЪДеЈ®е§ІдЄФе§НжЭВзЪДйАВеЇФжАІеЕНзЦЂпЉЙ
еЫЊ2 пЉИ1пЉЙClone ID:жѓПзІНеЕЛйЪЖзЉЦеПЈпЉМдїО0еЉАеІЛжМЙй°ЇеЇПзЉЦеПЈгАВе¶ВжЬЙжЬ™еЗЇзО∞зЪДзЉЦеПЈпЉМи°®з§Їиѓ•еЕЛйЪЖдЄЇout-of-frameжИЦдЄ≠йЧіжЬЙStop CondonзЪДпЉМйАЪеЄЄиЃ§дЄЇжШѓдЄНи°®иЊЊзЪДеЕЛйЪЖпЉМеЬ®жХ∞жНЃеИЖжЮРжЧґдЄАиИђдЉЪ襀ињЗжї§жОЙ пЉИ2пЉЙClone Count:жѓПзІНеЕЛйЪЖзЪДжАїжХ∞ пЉИ3пЉЙClone Fraction: и°®з§ЇжѓПзІНеЕЛйЪЖеН†жАїTCRеЕЛйЪЖзІНз±їзЪДйҐСзОЗзЪДзЩЊеИЖжѓФ пЉИ4пЉЙCDR3 Gene Sequence: CDR3ж†ЄйЕЄеЇПеИЧ пЉИ5пЉЙCDR3 AA Sequence: CDR3ж∞®еЯЇйЕЄеЇПеИЧ пЉИ6пЉЙV/D/J Segment: VDJеЯЇеЫ†зЙЗжЃµID

зђђдЇМйГ®еИЖпЉЪеЕНзЦЂзїДжµЛеЇПеИЖжЮР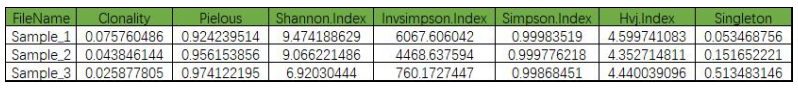 еЫЊ3
еЫЊ3
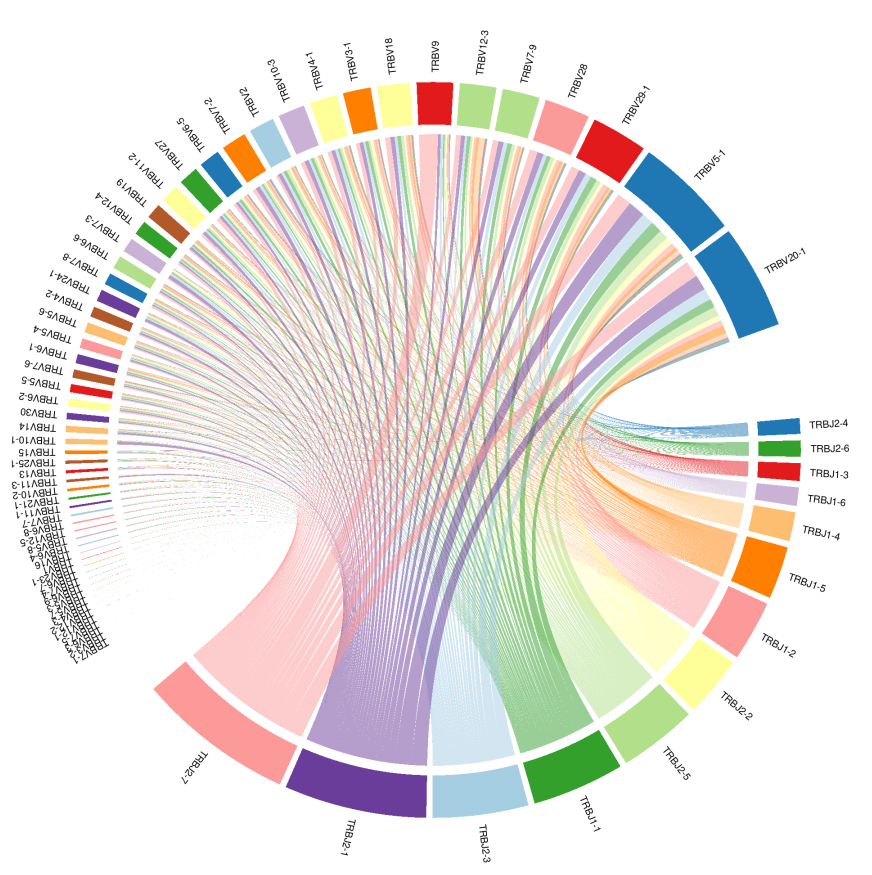 еЫЊ4
еЫЊ4V -J еЯЇеЫ†зЙЗжЃµзїДеРИеЫЊдљУзО∞зЪДжШѓеРДдЄ™ж†ЈжЬђзЪДV-JеЯЇеЫ†зЙЗжЃµдљњзФ®йҐСзОЗеТМзїДеРИйҐСзОЗпЉМеПѓиѓіжШОеЕНзЦЂз≥їзїЯе§Ъж†ЈжАІгАВе¶ВеЫЊжЙАз§ЇпЉМжѓПдЄАдЄ™иЙ≤еЭЧдї£и°®дЄАдЄ™еЯЇеЫ†зЙЗжЃµпЉМдЄКжЃµдЄЇ V зЙЗжЃµпЉМдЄЛжЃµдЄЇ J зЙЗжЃµпЉМиЙ≤еЭЧиґКеЃљдї£и°®ињЩдЄ™зЙЗжЃµдљњзФ®йҐСзОЗиґКйЂШпЉМињЮзЇњиґКе§ЪпЉМи°®з§ЇињЩзІН V-J зїДеРИйҐСзОЗиґКйЂШгАВ
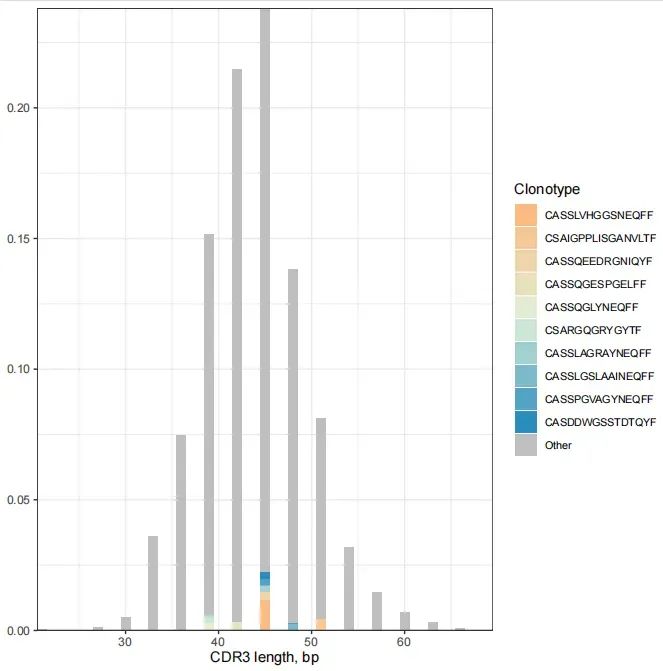
еЫЊ5
иЙЊж≤РиТљжПРдЊЫзЪДеРДдЄ™ж†ЈжЬђзЪДTop10 CDR3еЇПеИЧйХњеЇ¶еИЖеЄГжЯ±зКґеЫЊжМЙзЕІйҐСзОЗйАЙеПЦTop10дї•еПКеЕґдїЦпЉИOtherпЉЙеЕЛйЪЖпЉИдї•CDR3зЪДж∞®еЯЇйЕЄеЇПи°®з§ЇпЉЙзЪДCDR3ж†ЄйЕЄйХњеЇ¶еИЖеЄГпЉМXиљідЄЇCDR3ж†ЄйЕЄйХњеЇ¶пЉМYиљіжШѓйҐСзОЗпЉМдЄНеРМйҐЬиЙ≤и°®з§ЇдЄНеРМеЕЛйЪЖпЉМељ©иЙ≤и°®з§ЇTop 10еЕЛйЪЖпЉМзБ∞иЙ≤зЪДи°®з§ЇйЩ§Top 10дї•е§ЦзЪДеЕґдїЦеЕЛйЪЖгАВ
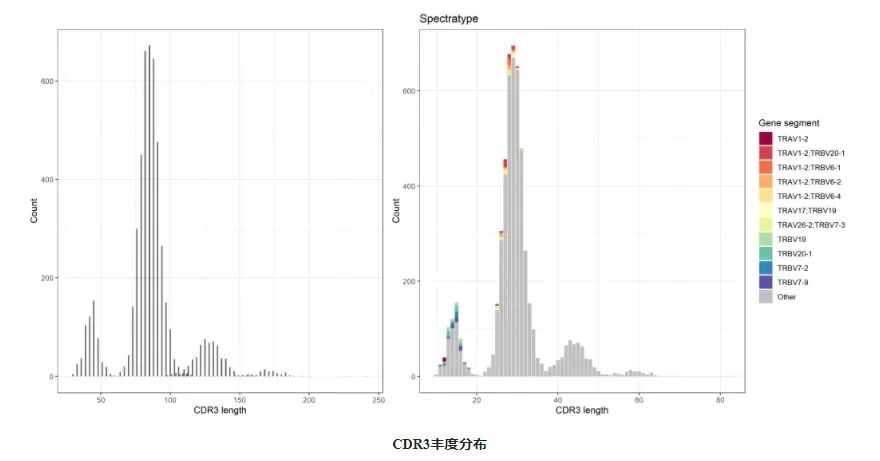
еЫЊ6
CDR3зФ±VгАБпЉИDпЉЙгАБJдЄЙдЄ™еЯЇеЫ†зЉЦз†БпЉМеЕЈжЬЙйЂШеПѓеПШжАІпЉМжШѓеЖ≥еЃЪT/BзїЖиГЮеЕЛйЪЖз±їеЮЛзЪДеМЇеЯЯпЉМеРМжЧґеЬ®жКЧеОЯиѓЖеИЂдЄ≠иµЈеЕ≥йФЃдљЬзФ®гАВиЗ™зДґзКґжАБдЄЛпЉМCDR3зЪДйХњеЇ¶еИЖеЄГеСИж≠£жАБеИЖеЄГгАВе¶ВеЫЊжЙАз§ЇпЉМйАЪињЗе∞ЖCDR3еЇПеИЧжМЙзЕІдЄАеЃЪзЪДйХњеЇ¶пЉИж≠§е§ДйАЙеПЦK=3пЉЙињЫи°МжЙУжЦ≠пЉМеПѓиОЈеЊЧkmerзїЯиЃ°зїУжЮЬпЉМжЬЙжК•йБУзІ∞еПѓдї•ж†єжНЃеЕґйЩНзїізїУжЮЬеѓєдЄНеРМзЪДиВњзШ§жЧґжЬЯињЫи°МеИЖеЮЛгАВ
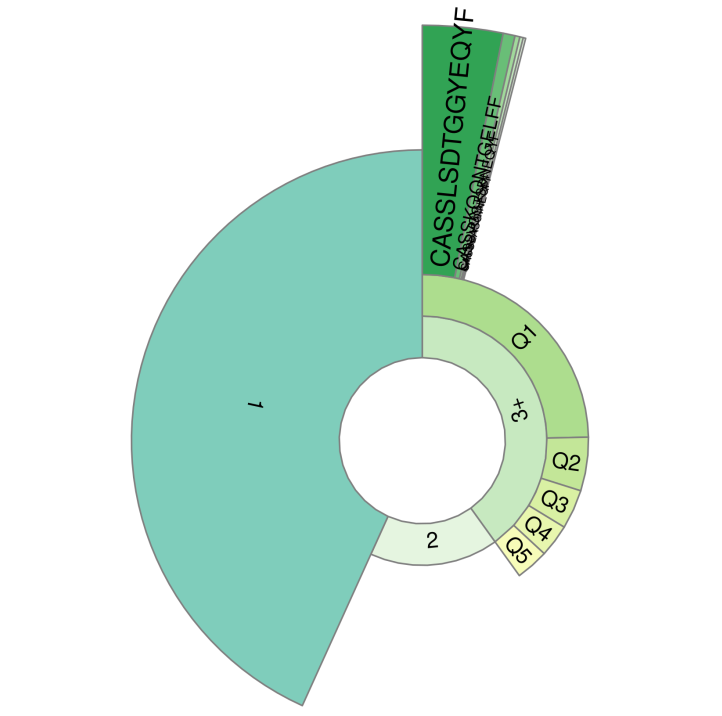 еЫЊ7
еЫЊ7еЕЛйЪЖжАІиЬЧзЙЫеЫЊеПѓеСИзО∞еЕЛйЪЖйҐСзОЗеИЖз±їгАВ
дї•TCRќ≤дЄЇдЊЛпЉЪ
зђђдЄАе±ВеМЕжЛђвАЬ1вАЭгАБвАЬ2вАЭеТМвАЬ3+вАЭпЉМеИЖеИЂи°®з§ЇжµЛеЇПжµЛеИ∞1жђ°гАБ2жђ°еТМ3жђ°дї•дЄКзЪДTCRеЕЛйЪЖеЇПеИЧзЪДзЩЊеИЖжѓФгАВвАЬ1вАЭеТМвАЬ2вАЭжЙАеН†зЪДжѓФдЊЛжШѓйЭЮеЄЄйЗНи¶БзЪДпЉМеЕґдЄОеИЭеІЛTзїЖиГЮжБѓжБѓзЫЄеЕ≥пЉМиГљељ±еУНTзїЖиГЮе§Ъж†ЈжАІзЪДиЃ°зЃЧгАВ
зђђдЇМе±ВзЪДQ1-Q5и°®з§ЇжѓП20%зЪДTCRеЕЛйЪЖеЇПеИЧжЙАеН†зЪДжѓФдЊЛпЉИTCRеЕЛйЪЖйҐСзОЗзФ±йЂШеИ∞дљОжОТеЇПпЉЙпЉМињЩиГљзЫіиІВеПНжШ†TCRеЕЛйЪЖжАІгАВ
зђђдЄЙе±ВжШѓеЙНtop5зІНTCRеЕЛйЪЖзЪДCDR3еМЇеЯЯзЪДж∞®еЯЇйЕЄеЇПеИЧгАВиѓ•е±ВеМЇеЯЯиґКеЃљпЉМиѓіжШОжЯРTCRеЕЛйЪЖжАІеҐЮжЃЦиґКйЂШгАВ
зђђдЄЙйГ®еИЖпЉЪеЕНзЦЂзїДжµЛеЇПеЇФзФ®вАФвАФеЕЛйЪЖжАІиЬЧзЙЫеЫЊ
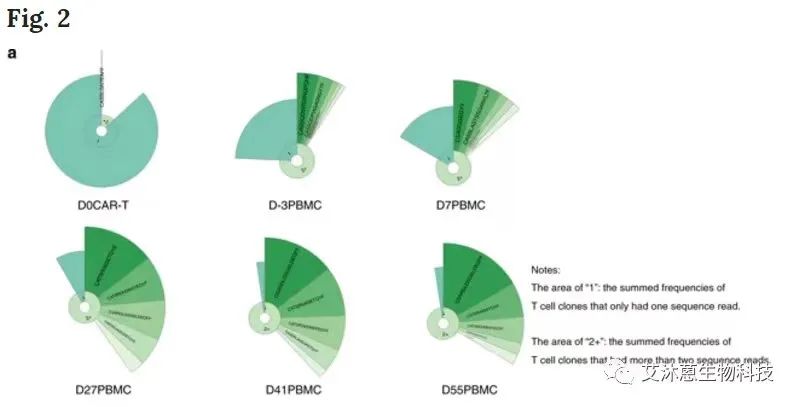
еЬ®иЙЊж≤РиТљеРИдљЬеПСи°®зЪДгАКQuantitative characterization of T-cell repertoire alteration in Chinese patients with B-cell acute lymphocyte leukemia after CAR-T therapyгАЛпЉМйАЪињЗImmuHub¬Ѓ¬†TCRжµЛеЇПпЉМеѓєдЄ≠еЫљзЩљи°АзЧЕжВ£иАЕCAR-Tж≤їзЦЧеРОTзїЖиГЮзїДеЇУпЉИTCRпЉЙеПШеМЦињЫи°Мз†Фз©ґгАВе¶ВдЄЛеЫЊжЙАз§ЇпЉМйАЪињЗTCRжµЛеЇПеѓєB-ALLжВ£иАЕCAR-Tж≤їзЦЧеЙНеТМж≤їзЦЧеРОе§ЪдЄ™жЧґйЧізВєзЪДе§ЦеС®и°АеТМй™®йЂУж†ЈжЬђињЫи°Мз†Фз©ґгАВиЬЧзЙЫеЫЊзїУжЮЬи°®жШОзїПињЗCAR-Tж≤їзЦЧеРОпЉМжВ£иАЕзЪДе§ЦеС®и°АеТМй™®йЂУдЄ≠еЭЗдЉЪеЗЇзО∞жШОжШЊзЪДTзїЖиГЮеЕЛйЪЖжАІеҐЮжЃЦгАВ
еЫЊ8пЉИеЫЊзЙЗжЭ•жЇРпЉЪhttps://www.nature.comпЉЙ
¬† ¬† ¬† ¬†еПВиАГжЦЗзМЃпЉЪ
        Wang X, Hu Y, Liu X, Yu J, Xu P, Wei G, Jin C, Wu W, Fu H, Ding L, Ni F, Zhang H, Liang Z, Wang B, Li X, Wei C, Deng Y, Shi J, Xiao L, Wu Z, Sun T, Huang H. Quantitative characterization of T-cell repertoire alteration in Chinese patients with B-cell acute lymphocyte leukemia after CAR-T therapy. Bone Marrow Transplant. 2019 Dec;54(12):2072-2080. doi: 10.1038/s41409-019-0625-y. Epub 2019 Aug 5. PMID: 31383996.
 
еЬ®еРИдљЬеПСи°®зЪДгАКThe Relationship Between Convergent IGH Signatures and Severity of COVID-19 Patients by Next-Generation Sequencing of B-Cell RepertoireгАЛжЦ∞еЖ†з†Фз©ґдЄ≠пЉМйАЪињЗImmuHub¬Ѓ BCRжµЛеЇПеє≥еП∞пЉМеПСзО∞BзїЖиГЮеПѓдї•иѓЖеИЂжЦ∞еЮЛеЖ†зКґзЧЕжѓТпЉИSARS-CoV-2пЉЙеєґдЇІзФЯеЕЛйЪЖжАІеҐЮжЃЦгАВе¶ВдЄЛиЬЧзЙЫеЫЊеИЖжЮРзїУжЮЬжЙАз§ЇпЉМ襀зФ®жЭ•иѓДдЉ∞жВ£иАЕзЪДIGHеЇУеК®жАБеТМж≤їзЦЧжХИжЮЬгАВеЫЊдЄ≠жШЊз§ЇдЇЖIGHзЪДдЇТи°•еЖ≥еЃЪеМЇпЉИCDR3пЉЙзЪДеЙНдЇФдЄ™еЕЛйЪЖзЪДж∞®еЯЇйЕЄеЇПеИЧгАВдЄГеРНеН±йЗНжВ£иАЕ襀еИЖдЄЇжФєеЦДзїДеТМжБґеМЦзїДпЉМж≤їзЦЧ7姩еРОпЉМжФєеЦДзїДеЗЇзО∞еЕЛйЪЖжАІеҐЮжЃЦпЉМеЙН5дЄ™еЕЛйЪЖзЪДжАїйҐСзОЗеҐЮеК†пЉМзДґиАМпЉМеЬ®жБґеМЦзїДдЄ≠пЉМеЙН5дЄ™еЕЛйЪЖжАїйҐСзОЗеЗПе∞ПгАВ
 
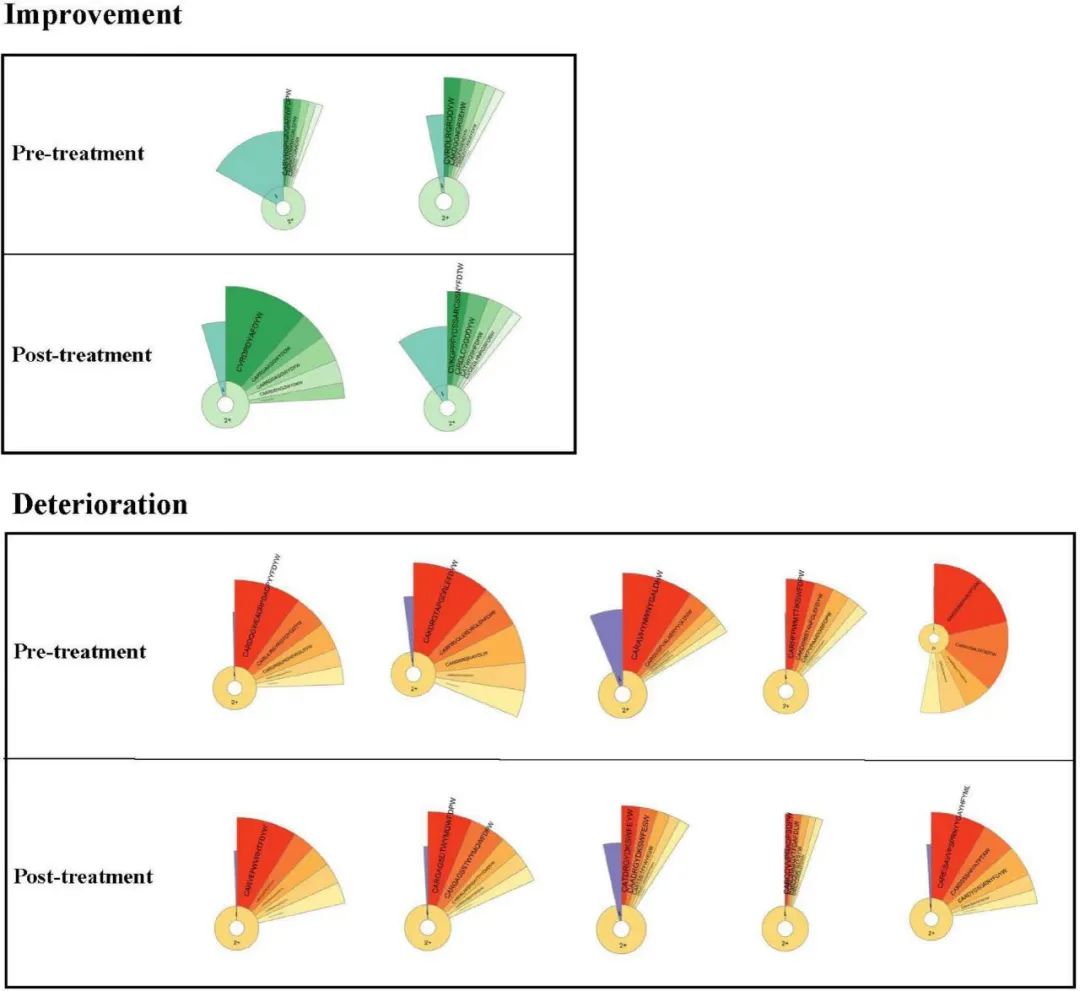 ¬†еЫЊ.¬†¬†IGH зЪДиЬЧзЙЫеЫЊгАВдЄГеРНжВ£иАЕеЬ®ж≤їзЦЧеРОеТМж≤їзЦЧеЙНзЪДиЬЧзЙЫеЫЊгАВжЙЗ嚥еМЇеЯЯжППзїШдЇЖзЫЄеЇФзЪДеЕЛйЪЖйҐСзОЗгАВеЫЊдЄ≠вАЬ1вАЭзЪДеМЇеЯЯдї£и°®жµЛеЇПжµЛеИ∞1жђ°зЪДйҐСзОЗгАВеЉІеЇ¶йЭҐзІѓиґКе§ІжДПеС≥зЭАзЪДеЕЛйЪЖйҐСзОЗиґКйЂШгАВеЫЊдЄ≠жШЊз§ЇдЇЖ IGH зЪДдЇТи°•еЖ≥еЃЪеМЇ (CDR3) зЪДеЙНдЇФдЄ™еЕЛйЪЖзЪДж∞®еЯЇйЕЄеЇПеИЧгАВ
¬†еЫЊ.¬†¬†IGH зЪДиЬЧзЙЫеЫЊгАВдЄГеРНжВ£иАЕеЬ®ж≤їзЦЧеРОеТМж≤їзЦЧеЙНзЪДиЬЧзЙЫеЫЊгАВжЙЗ嚥еМЇеЯЯжППзїШдЇЖзЫЄеЇФзЪДеЕЛйЪЖйҐСзОЗгАВеЫЊдЄ≠вАЬ1вАЭзЪДеМЇеЯЯдї£и°®жµЛеЇПжµЛеИ∞1жђ°зЪДйҐСзОЗгАВеЉІеЇ¶йЭҐзІѓиґКе§ІжДПеС≥зЭАзЪДеЕЛйЪЖйҐСзОЗиґКйЂШгАВеЫЊдЄ≠жШЊз§ЇдЇЖ IGH зЪДдЇТи°•еЖ≥еЃЪеМЇ (CDR3) зЪДеЙНдЇФдЄ™еЕЛйЪЖзЪДж∞®еЯЇйЕЄеЇПеИЧгАВ
еПВиАГжЦЗзМЃпЉЪ
Cai, Hongliu et al. вАЬThe Relationship Between Convergent IGH Signatures and Severity of COVID-19 Patients by Next-Generation Sequencing of B-Cell Repertoire.вАЭ¬†Frontiers in microbiology¬†vol. 12 833054. 9 Feb. 2022, doi:10.3389/fmicb.2021.833054
жАїзїУ
иЙЊж≤РиТљжПРдЊЫдї•дЄКдЄЙзІНжЈ±еЇ¶еИЖжЮРдљЬдЄЇж†ЗеЗЖеИЖжЮРжК•еСКгАВиЛ•жЬЙеЕґдїЦжЫіжЈ±е±Вжђ°зЪДйЬАж±ВпЉМиЙЊж≤РиТљдЄНдїЕиГљжПРдЊЫдЇМеНБдљЩзІНжЈ±еЇ¶еИЖжЮРпЉМињШеПѓдї•еЃЮзО∞еРДзІНдЄ™жАІеМЦйЬАж±ВеИЖжЮРгАВдљ†еПѓдї•дїАдєИйГљдЄНзЫЄдњ°пЉМдљЖжШѓдЄАеЃЪи¶БзЫЄдњ°иЙЊж≤РиТљзЪДжКАжЬѓгАВ
жЭ≠еЈЮиЙЊж≤РиТљзФЯзЙ©зІСжКАжЬЙйЩРеЕђеПЄзФ±зЊОеЫљиКЭеК†еУ•е§Іе≠¶зІСз†ФеЫҐйШЯеЫЮеЫљеИЫеКЮпЉМжШѓдЄАеЃґдЄУж≥®дЇОйАЪињЗиІ£з†БйАВеЇФжАІеЕНзЦЂз≥їзїЯжЭ•жФєеПШзЦЊзЧЕзЪДиѓКжЦ≠еТМж≤їзЦЧпЉМеєґиЗіеКЫдЇОжО®ињЫеЕНзЦЂй©±еК®еМїе≠¶йҐЖеЯЯеПСе±ХзЪДеЫљеЃґйЂШжЦ∞жКАжЬѓдЉБдЄЪгАВиЙЊж≤РиТљзЂЩеЬ®йАВеЇФжАІеЕНзЦЂз≥їзїЯз†Фз©ґзЪДеЙНж≤њпЉМиЗ™дЄїз†ФеПСзЪДеЕНзЦЂеМїе≠¶еє≥еП∞еПѓжП≠з§ЇеТМзњїиѓСйАВеЇФжАІеЕНзЦЂз≥їзїЯзЪДйБЧдЉ†еѓЖз†БпЉМеєґиГљеЇФзФ®дЇОзЩМзЧЗгАБиЗ™иЇЂеЕНзЦЂжАІзЦЊзЧЕгАБдЉ†жЯУжАІзЦЊзЧЕз≠ЙеЕНзЦЂдїЛеѓЉжАІзЦЊзЧЕзЪДиѓКжЦ≠гАБзЫСжµЛеТМж≤їзЦЧдЄ≠гАВ

