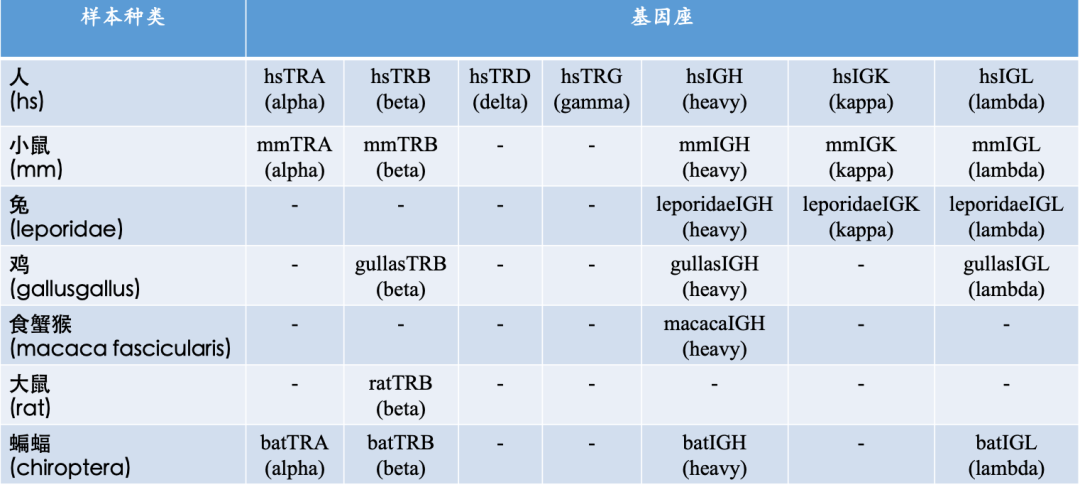
导读:
近日,艾沐蒽助力遵义医科大学姚新生教授团队发表了国际首篇有关蝙蝠免疫组T细胞受体(TCR)测序的研究论文“New insights into the germline genes and CDR3 repertoire of the TCRβ chain in Chiroptera”。
姚教授团队是国内最早探索免疫组学测序的先行者,于2007年便发表了国内第一篇免疫组测序的文章。艾沐蒽此次很荣幸能与姚教授团队合作,结合艾沐蒽在免疫组学建库测序技术上的优势,助力解析蝙蝠免疫组基因测序成果发表,未来双方将在免疫组学领域建立更多深入合作探讨,促进免疫组学发展。

引言:蝙蝠被认为是许多病毒的天然宿主,它们独特的免疫系统使它们能够与这些病毒共存,而不经常表现出疾病症状。然而,由于缺乏能够处理蝙蝠T细胞受体(TCR)序列的数据库或工具,目前对蝙蝠适应性免疫系统的了解有限。
方法:采用同源基因和重组信号序列(RSSs)扫描方法对3种蝙蝠进行胚系基因注释。然后利用保守的C基因构建了中菊头蝠的TCRβ链受体文库。Bats的TCRβ数据将使用MiXCR和构建的参考库进行分析。
结果:根据注释结果,我们发现苍白矛吻蝠TRBV12家族有37个成员,超过了马铁菊头蝠TRBV基因的总数。每个中菊头蝠样本中独特的TCRβ链受体序列的平均数量达到24,904个。
讨论:三种蝙蝠中TRBV基因分布的明显差异可能对TCR库的多样性产生直接影响,保守氨基酸的存在表明蝙蝠中T细胞对抗原的识别受MHC限制。蝙蝠的TCRβ库是通过V-D- J – C基因的重排形成的,D-J/V-D基因的缺失和插入导致高度多样性。
蝙蝠属于翼手目,是世界上第二大的哺乳动物目(1)。尽管携带了许多致命的病毒,蝙蝠却没有出现严重病症(2-5),这使得人们对蝙蝠免疫系统与其他哺乳动物免疫系统之间的差异越来越感兴趣。
随着现在(NGS)测序技术促进了高通量测序(HTS)对不同物种的基因组分析。蝙蝠1k计划和脊椎动物基因组计划(VGP)也产生了高质量的蝙蝠基因组数据,这提高了我们对蝙蝠免疫系统的认识,特别是它们对病毒感染的反应(14,15)。但是蝙蝠T/B细胞受体胚系基因的注释仍然有限。
TCR/BCR库包括个体循环系统中的所有功能T或B细胞,每个细胞都有自己的抗原特异性受体。基因库的深度测序是研究适应性免疫系统、评估个体健康状况、开发抗体以及检测和治疗靶向疾病的常用方法(16)。各种工具,如IMGT/HighV-QUEST, TRUST, MiXCR,已被用于处理TCR/BCR序列(17-19)。作者之前的工作涉及到R. ferrumequinum TRB位点的注释,表明蝙蝠的适应性免疫反应与人类和小鼠相似,其TCR是由具有高度多样性的胚系基因重排而来。
在本研究中,作者将注释扩展到了包括P. discolor和P. pipistrellus的TRB位点,并比较了这三种蝙蝠的TRB位点。对不同种类蝙蝠的TRBC Exon1序列进行比对,利用5 ‘ race技术,开发引物扩增R. affinis TCR β链CDR3序列。本研究为研究翼手目遗传进化和适应性免疫反应提供了重要的理论基础、研究方法和可比性数据库。
蝙蝠样本选择了三种R. affinis(中菊头蝠)作为实验模型,因为它们与目标物种R.ferrumequinum (马铁菊头蝠)具有很高的同源性。
采用3只5月龄BALB/c小鼠脾脏组织。
此外,作者还使用了3个人的5ml外周血样本。

通过杭州艾沐蒽运用ImmuHub® 技术对R. affinis(中菊头蝠)TCR-β链进行文库构建和高通量测序。
小鼠脾组织采用5’race法构建TCR-β库,人外周血单个核细胞(PBMC)采用多重PCR法构建TCR-β库。
然后对测序结果使用MiXCR软件(Version 3.0.13)分析了蝙蝠、人类和小鼠的测序结果
研
究
结
果
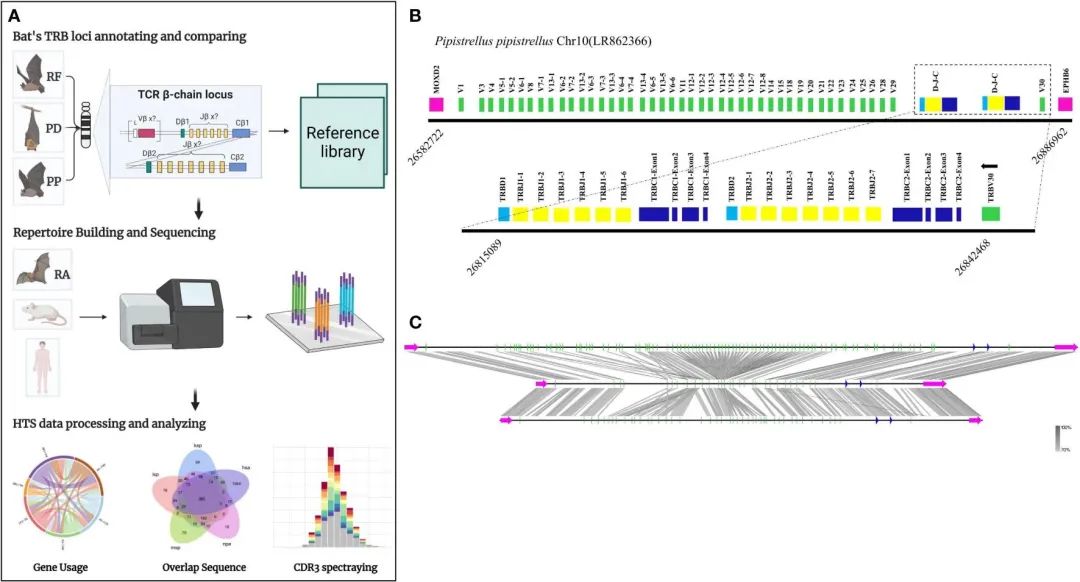
FIGURE 1 The TRB loci annotation and correlation analysis of three bat species.
图1A展示了本研究的实验流程。其中包括通过检测位于TRB位点边界的两个基因MOXD2和EPHB6来识别基因组中的TRB位点。使用两种基因注释方法IMGT-LIGMotif(21)和12/23 RSS(Recombination Signal Sequence,重组信号序列)扫描对两个基因之间的区域进行注释。
另外还使用Easyfig (Version 2.2.5)比较三种蝙蝠注释的TRB位点的同源性,识别阈值为70%(图1C)。
表1显示在P. discolor 中发现了TRB的位置第10染色体(NC_040912:85517683-85943353)、R. ferrumequinum(马铁菊头蝠)的TRB位置在第26染色体(NC_046309:7911840-8171343)、P.pipstrellus(伏翼)的TRB位置在第10染色体(LR862366.1:26582722-26886962)。这三个物种的TRB位点均遵循经典的哺乳动物结构,但在长度分布和取向上存在差异,如图1B和补充图1所示。
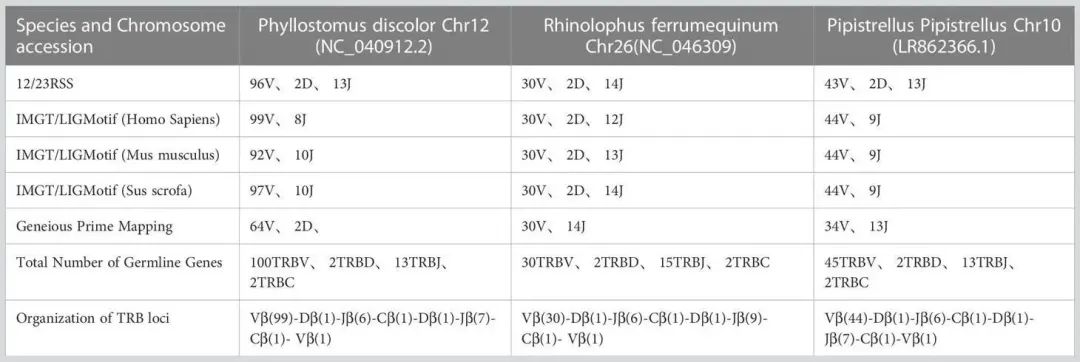
TABLE 1 Number of germline genes retrived by homologous genes and RSSs method.
作者通过IMGT-LIGMotif方法选取了人类(灵长类)、小鼠(啮齿动物)和猪(偶蹄目动物)三种不同主体的代表性动物,获取IMGT中所有已知物种的TRBV基因,以确保不因进化分歧而遗漏信息。使用geneious Prime (Version 2022.2.1)将这些基因定位到三种蝙蝠的TRB位点。
12/23 RSS扫描方法包括筛选三个蝙蝠TRB位点内的所有RSS基序,并在RSS的上游和下游寻找V/D/J基因。
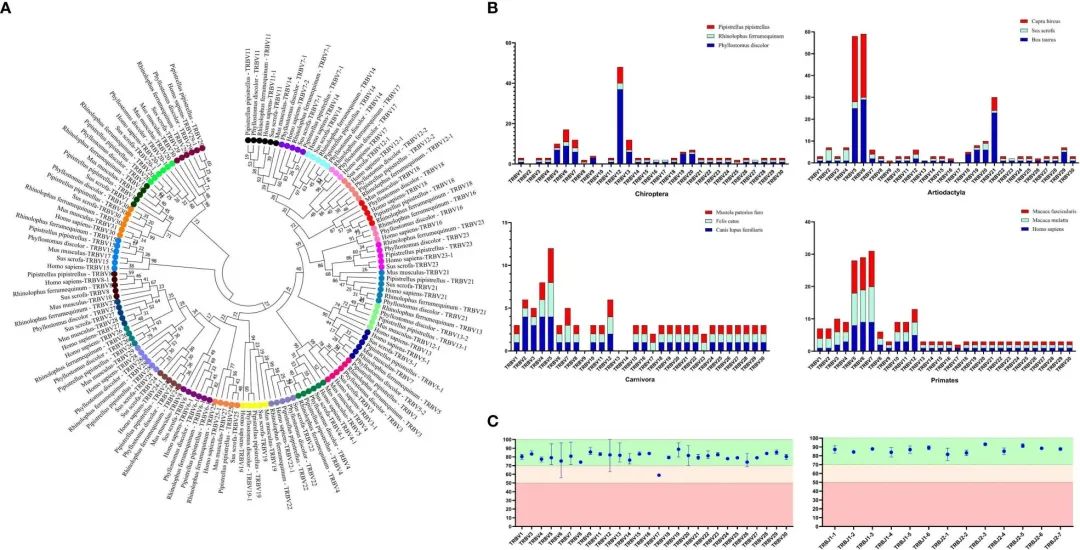
FIGURE 2 Analysis of TRBV gene homology in different bat species, as well as in human, mice, and pigs.
作者为了将V基因划分为家族,设置了75%核苷酸同一性的阈值,作者为了验证家族同源性,利用MEGA7 (version 7.0.26)的Neighbor-Joining方法构建了TRBV核苷酸序列的系统进化树。从IMGT-GeneDB (https://www.imgt.org/ genedb/)检索人、小鼠和猪的TRBV基因,只选择功能基因和阅读框架区 ,每个家族一个基因。补充表1列出了分析中使用的接入号。比较了TRBV基因家族在属于四个不同种目(偶蹄目、食肉动物和灵长类动物)的12种哺乳动物物种中的分布,以了解V基因的进化方向(图2A)。D、J和C基因是根据它们在基因座中的位置来命名的。
通过分析结果表明,在R. ferrumequinum中鉴定出100个TRBV基因(含2个假基因),在R. ferrumequinum中鉴定出30个TRBV基因,在P. pipistrellus中鉴定出45个TRBV基因。
利用TRBV基因的核苷酸序列构建了系统进化树,结果表明所有基因可以彼此同源,也可以在猪和人类中发现的基因同源。在蝙蝠中,不存在单独聚类的情况(图2A)。种系基因数量的主要差异是由于V基因家族的内部重复和缺失。Chiroptera(翼手目)和 Artiodactyla(偶蹄目)的进化方向更为相似,如图2B所示。除了TRBV17家族外,三种蝙蝠的TRBV和TRBJ基因家族的同源性超过70%(图2C),作者鉴定了一个特殊的TRBV12家族,该家族包含37个成员,接近于P. pipistrellus的几乎所有TRBV基因,甚至超过了R. ferrumequinum的TRBV基因总数。
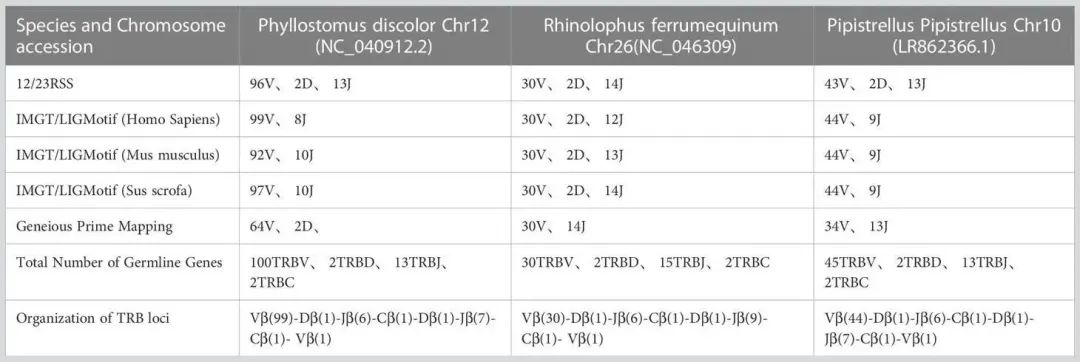
TABLE 2 MiXCR outputted sequence statistics from a total of nine samples, including bats, humans, and mice.
使用MiXCR软件(Version 3.0.13)分析了蝙蝠、人类和小鼠的测序结果。基于注释基因构建的文库,利用MiXCR对蝙蝠的测序结果进行组装和呈现。九个样本的输出数据如表2和补充表6所示。为了保证结果的质量,我们检查了bat1样本含有25个TRBV基因和14个TRBJ基因的序列(图4E)。
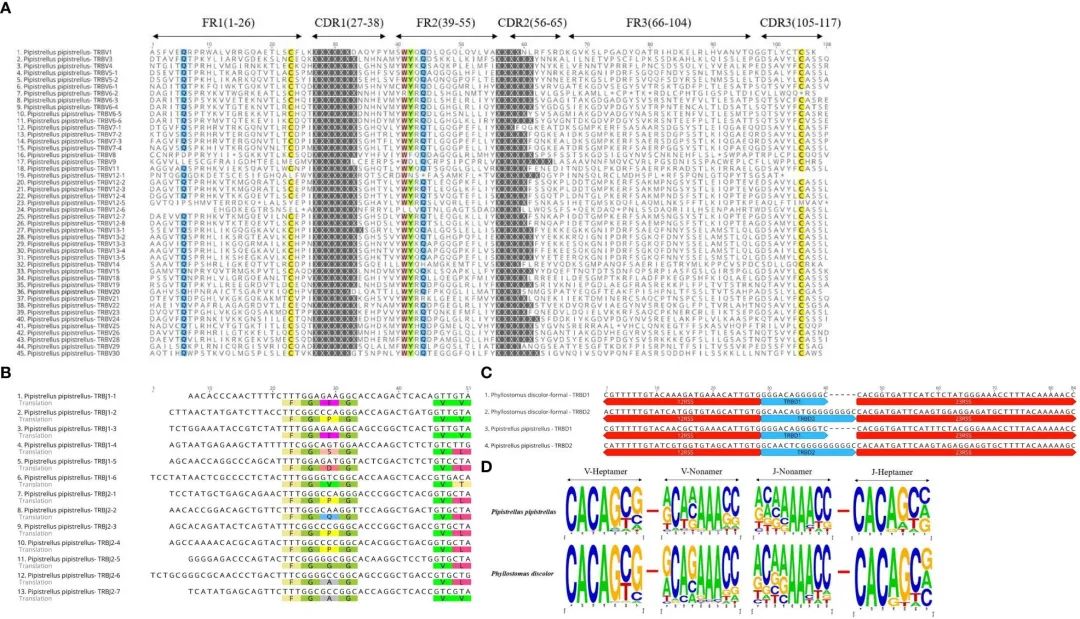
FIGURE 3 Annotated Germline Genes Display.
在TRBV基因的多序列比对(Multiple Sequence Alignment, MSA)中,我们发现了23Cys、41Trp、89Leu和104Cys等几个保守位点,Gln位于第6位,Tyr位于第42位。第44位的Gln在P. pipistrellus中也高度保守。此外,大多数TRBV基因在末端都含有CASS基序(图3A和补充图2A)。
R. ferrumequinum(7%)和P. pipistrellus(11%)相比,P. discolor具有更高的胚系基因数量和更多的基因重复,导致假基因数量(17%)更多。而 P. discolor的功能基因(V和J基因)数量为97个,超过了R. ferrumequinum和P. pipistrellus(补充表2)。
像灵长类动物和食肉动物一样,翼手目包含两个根据染色体位置命名的D-J-C簇。这三种蝙蝠在每一簇基因数量上的唯一区别是第二簇J基因的数量。蝙蝠的TRBJ基因除R的TRBJ2-3外均含有保守的FGNG基序。(图3B和补充图2B)。
此外,所有蝙蝠TRBD基因均为鸟嘌呤富集序列,上下游分别为23RSS和12RSS(图3C)。
蝙蝠与其他哺乳动物的RSS没有显著差异,七聚体的前四个位点(CACA)最为保守,三个连续的腺苷位于非聚体的中间位置(图3D和补充图3)。
作者创建了一个MiXCR参考库,用于使用核苷酸序列和氨基酸序列MSA结果进行HTS数据分析。为了组织三种蝙蝠的胚系基因家族,用等位基因命名法将它们组合成一个单一的家族(补充表3)。

FIGURE 4 Homology analysis of R. affinis samples and annotated bat species.
为了进一步研究对TCRβ基因库进行了构建、测序和分析。通过对3只实验蝙蝠的Cytb测序结果进行分析,平均序列长度为488个碱基对,序列一致性为98.5%
发现19个TRBC Exon1的核苷酸序列同源性在77.66% ~ 98.9%之间,有几个完全保守的区域适合构建库和测序,如图4A所示。
为了探究蝙蝠物种分化的程度,作者对三种实验蝙蝠R.affinis、R. ferrumequinum 和P. pipistrellus进行了比较,结果表明,R. ferrumequinum和R.affinis同源性(87.9%)高于P. pipistrellus(74.6%)。R.affinis与P. discolor、R. ferrumequinum和P. pipistrellus之间的TRBC核苷酸片段的同源性,分别为77.1%, 93.8%和 75.0%(图4C)。对实验样品的测序结果进行分类后,作者发现R. affinis的大部分输出序列属于R. ferrumequinum(图4D)。
这些比较结果进一步表明,R. affinis的序列与R. ferrumequinum的序列更为相似。然后,我们从Bat1样本中导出25 V和14 J基因序列,证明了该库的完整性(图4E)。
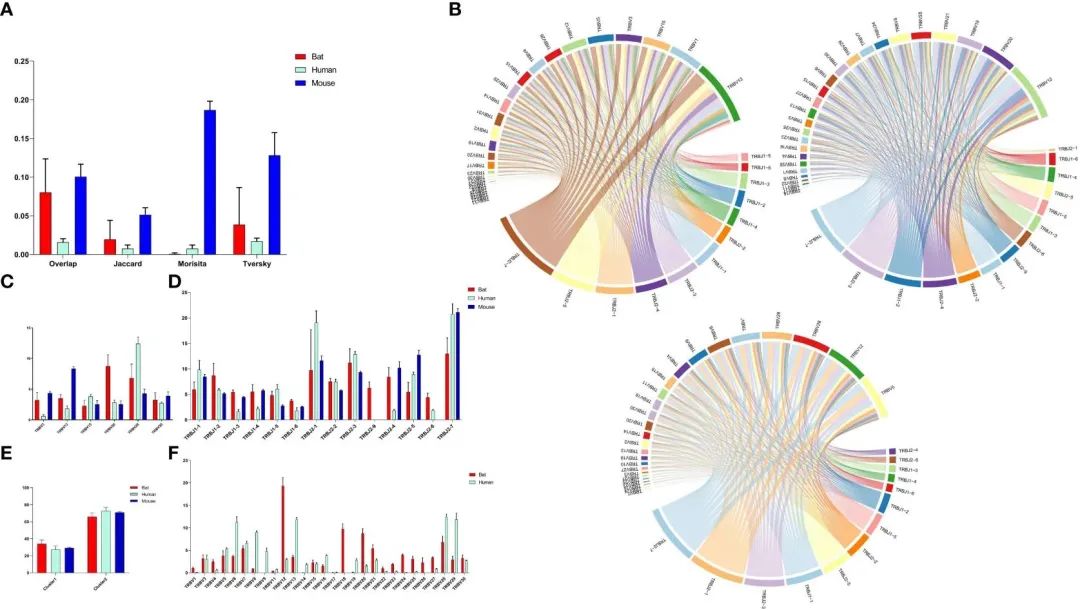
FIGURE 5 Comparative Analysis of TCR b Chain Repertoire Genes and Motif Usage in Bats, Humans, and Mice.
为了比较蝙蝠、人类和小鼠CDR3的基因库,作者通过(重叠系数、Jaccard、Morisita和Tversky)四项指标评估了蝙蝠、人类和小鼠的CDR3库(图5A)。分析结果表明,蝙蝠CDR3谱变化较大,彼此之间差异显著,部分重叠系数略高于人类,但显著低于小鼠。
基于系统树的结果,作者分析了蝙蝠、人类和小鼠TRBV基因使用的同源性和V-J配对(图5B和补充图5)。有6个同源TRBV基因表达,其中TRBV15和TRBV30表达相似(图5C)。此外,我们比较了所有TRBV基因在蝙蝠和人类中的使用情况(图5F)。蝙蝠中最常用的TRBV基因是TRBV12、TRBV18和TRBV20,而在人类中,它们是TRBV13、TRBV28和TRBV29。值得注意的是,TRBV9和TRBV19在蝙蝠样本中要么不表达,要么低表达。此外,仅有P. discolor的TRBV9组中四个TRBV9成员都是假基因。在这三个物种中,除了蝙蝠的TRBJ2-8(假基因)和小鼠的TRBJ2-6(假基因)外,所有功能TRBJ基因都得到了表达(图5D)。通过比较TRBJ分析,作者发现TRBJ2-1、TRBJ2-3和TRBJ2-7在蝙蝠、人类和小鼠中同时出现高使用率。此外,在三个物种中使用的大部分J基因来自第二个D-J-C簇(图5E)。这一发现表明发生基因重排过程与距离有关。
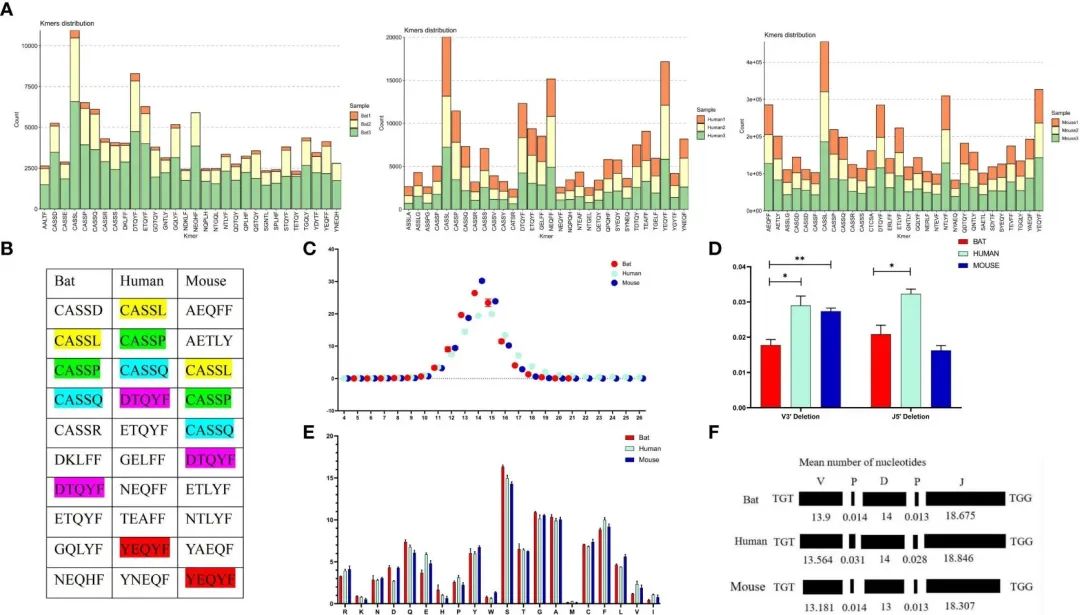
FIGURE 6 Comparative analysis of the CDR3 region features of the TCRb chain repertoire in bat, human, and mouse is presented in this figure.
通过分析了CDR3区域基序(5个氨基酸),发现前10个高频基序中有4个在蝙蝠、人类和小鼠中共用,其中大多数是CASSN基序(图6A, B)。这表明三个物种中参与免疫反应的细胞群可能是相似的。
蝙蝠和小鼠以14个氨基酸长度为中心、人类CDR3呈现以15个氨基酸长度为中心的钟形分布(图6C)。
作者评估了CDR3区域核苷酸插入和缺失引起的长度效应,发现人类和小鼠在V’3端有相似的缺失,而蝙蝠和小鼠在V’3端和J基因5’端有相似的插入(图6D, E)。最后,我们观察到CDR3区域的氨基酸是一致的,在所有三个物种中都观察到S、G和A的高频(图6F)。
1.三种蝙蝠中TRBV基因分布的明显差异可能对TCR库的多样性产生直接影响,保守氨基酸的存在表明蝙蝠中t细胞对抗原的识别受MHC限制。
2. 蝙蝠的TCRβ库是通过V-D- J – C基因的重排形成的,D-J/V-D基因的缺失和插入导致高度多样性。
总结:蝙蝠的T细胞免疫应答依靠细胞表面的T细胞受体(TCR)对抗原进行特异性识别及应答。本研究对3种蝙蝠(马铁菊头蝠、苍白矛吻蝠、伏翼)TCR重链胚系基因进行了详细注释。对于TCR ,共注释了174个V基因片段,分为30个家族;2个D基因家族;2个J基因家族。所有的胚系基因根据胚系基因结构信息区分了不同的结构域,并以此建立了蝙蝠TCR参考数据库。
进一步,本研究完成了中华菊头蝠TCR组库测序,利用参考数据库进行了TCR组库分析(多样性组成、克隆分布频率、CDR3长度分布及构成、V(D)J连接区插入和剪切(N/P)、V-J配对及特征、CDR3氨基酸取用分布),并与人及小鼠TCR组库的特征进行比对。
本研究首次高通量地解析了蝙蝠的TCR组库,其展现出高度多样性,表明蝙蝠适应性免疫系统可以特异性识别种类庞大的抗原;蝙蝠TCR 序列在结构域特定位点具有高度保守氨基酸,和TCR识别存在MHC限制性相关;蝙蝠胚系基因末端RSS序列保守,说明蝙蝠TCR重排机制与人及小鼠类似,是由酶介导的剪切及连接;但蝙蝠家族取用与人及小鼠存在差异,提示其免疫系统识别抗原与人及小鼠存在不同,这或许是其能够与病毒共存的原因。
Zhou H, Li J, Zhou D, Wu Y, Wang X, Zhou J, Ma Q, Yao X and Ma L (2023) New insights into the germline genes and CDR3 repertoire of the TCRβ chain in Chiroptera.Front. Immunol. 14:1147859.doi: 10.3389/fimmu.2023.1147859
什么是ImmuHub®技术?
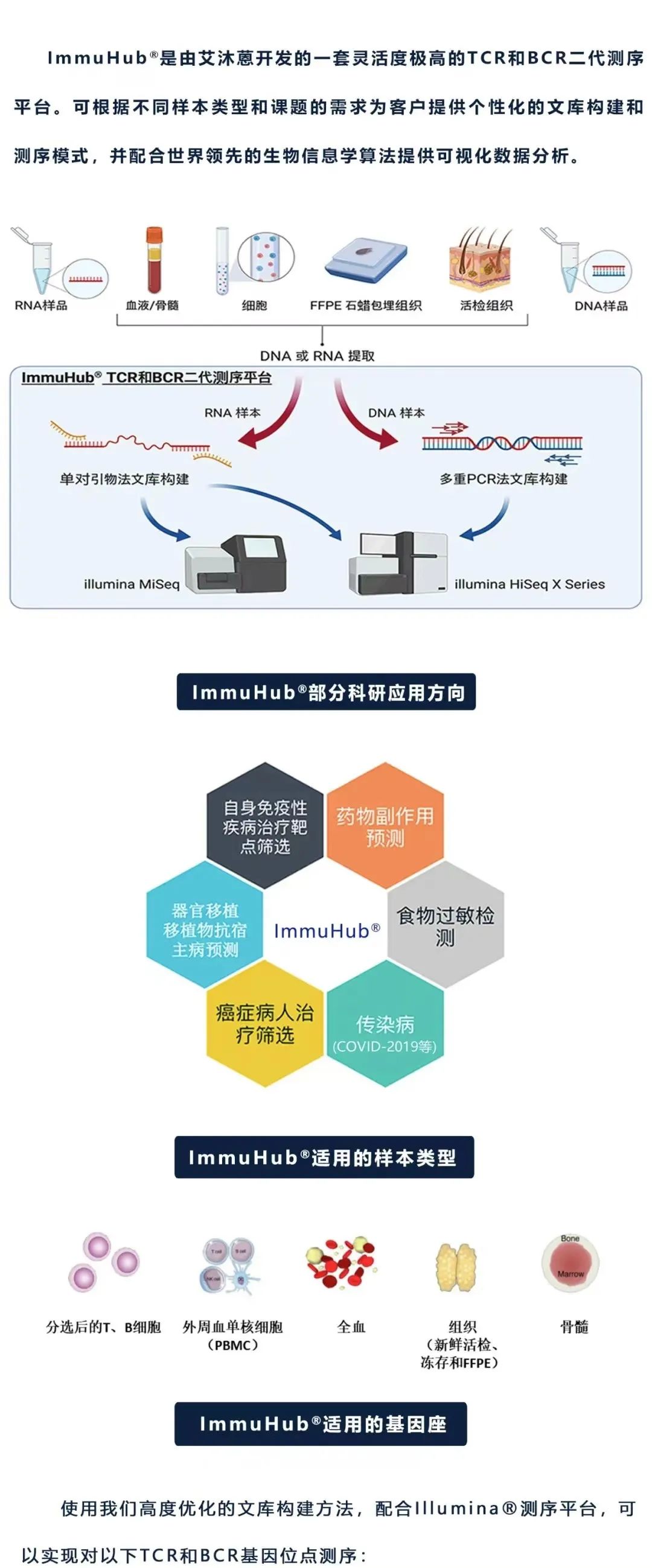
杭州艾沐蒽生物科技有限公司由美国芝加哥大学科研团队回国创办,是一家专注于通过解码适应性免疫系统来改变疾病的诊断和治疗,并致力于推进免疫驱动医学领域发展的国家高新技术企业。艾沐蒽站在适应性免疫系统研究的最前沿,自主研发的免疫医学平台可揭示和翻译适应性免疫系统的遗传密码,并能应用于癌症、自身免疫性疾病、传染性疾病等免疫介导性疾病的诊断、监测和治疗中。

