пЉЛ
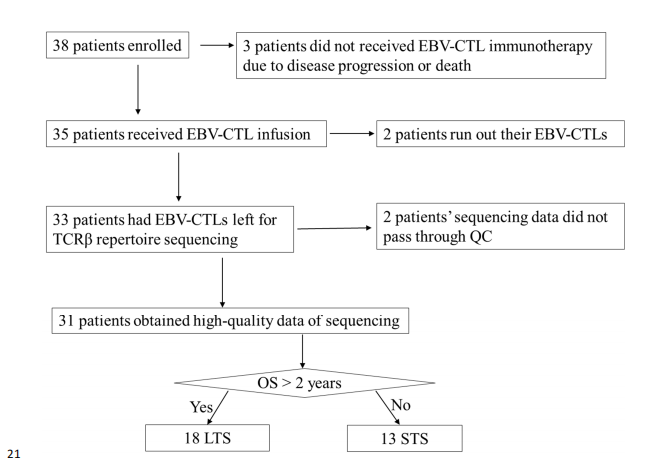
еЬ®ињЗеОїзЪДеНБеєідЄ≠пЉМињЗзїІжАІTзїЖиГЮзЦЧж≥ХеЬ®ж≤їзЦЧеРДзІНз±їеЮЛзЪДзЩМзЧЗжЦєйЭҐеПЦеЊЧдЇЖйАРж≠•еТМз®≥еЃЪзЪДињЫе±ХгАВдљЬиАЕеЬ®ињЫи°МдЄАй°єдЄіеЇКиѓХй™МNCT00690872дЄ≠пЉМиБФеРИеРЙи•њдїЦжї®еТМеН°йУВеМЦзЦЧж≤їзЦЧEBзЧЕжѓТ(EBV)йШ≥жАІйЉїеТљзЩМ(NPC)жВ£иАЕиЗ™дљУEBVжЙ©еҐЮзїЖиГЮжѓТжАІTжЈЛеЈізїЖиГЮ(CTL)гАВиЩљзДґжЬЙ62.9%зЪДиЊЊеИ∞2еєіжАїзФЯе≠ШзОЗпЉМдљЖиѓ•иѓХй™Мз°Ѓж≤°жЬЙеЬ®дЄАйГ®еИЖжВ£иАЕдЄ≠дЇІзФЯиѓ±еѓЉжКЧиВњзШ§еПНеЇФгАВеЫ†ж≠§пЉМдїНзДґињЂеИЗйЬАи¶БзЪДиѓДдЉ∞CTLдЇІзЙ©еТМйҐДжµЛдЄіеЇКеЕНзЦЂж≤їзЦЧжХИжЮЬзЪДеЯЇеЗЖпЉМдЇОжШѓдљЬиАЕйАЙжЛ©ињЫи°МдЇЖTзїЖиГЮеПЧдљУ(TCR)еЕ®еЇУжµЛеЇПпЉМжЭ•иѓДдЉ∞EBVжЙ©еҐЮзЪДиЊУж≥®еЃМжИРзЪДCTLдЇІзЙ©гАВдЄЇдЇЖжППзїШжХідљУзЪДеЇУжЩѓиІВпЉМдљЬиАЕйАЪињЗй¶ЩеЖЬзЖµиѓДдЉ∞дЇЖдЄ™дљУеЇУе§Ъж†ЈжАІпЉМеєґжѓФиЊГдЇЖжВ£иАЕйЧіCDR3зЫЄдЉЉжАІжЭ•дЉ∞иЃ°зФ±еЕ±еРМжКЧеОЯжЙ©еҐЮзЪДTзїЖиГЮгАВеИ©зФ®жЬАињСеЉАеПСзЪДдЄАзІНзІ∞дЄЇMotif AnalysisзЪДзФЯзЙ©дњ°жБѓе≠¶зЃЧж≥ХпЉМеѓєTCRќ≤ CDR3еЖЕдЄОCTLж≤їзЦЧйҐДеРОзЫЄеЕ≥зЪДзїУжЮДеМЇеЯЯињЫи°МдЇЖжЬЇеЩ®е≠¶дє†йҐДжµЛгАВ
зїУжЮЬи°®жШОпЉМеРЂжЬЙеЕ±еРМж†ЄењГеЯЇеЇПйЫЖзЪДдЄНеРМTCRеЇПеИЧдЄОEBVйШ≥жАІйЉїеТљзЩМпЉИNPCпЉЙзЪДCTLеЕНзЦЂж≤їзЦЧзЪДжЬЙеИ©еПНеЇФжЬЙеЕ≥гАВ
дЄЛйЭҐе∞±иЃ©жИСдїђдЇЖиІ£дЄАдЄЛпЉБ

пЉЛ
з†Фз©ґжЦєж≥Х
1.з†Фз©ґиЃЊиЃ°
2.TCRжЦЗеЇУжЮДеїЇеПКTCRќ≤жµЛеЇП
3.еОЯеІЛжХ∞жНЃпЉИRaw dataпЉЙе§ДзРЖ
4.е§Ъж†ЈжАІеИЖжЮР
5.зЫЄдЉЉжАІеИЖжЮР
6.Motif еИЖжЮР
7.зїЯиЃ°еИЖжЮР
пЉЛ
з†Фз©ґзїУжЮЬ
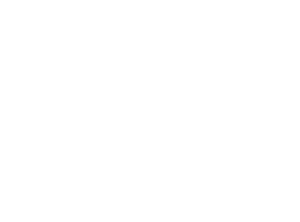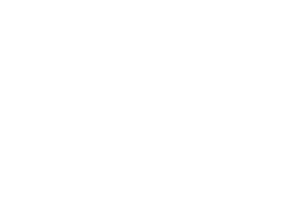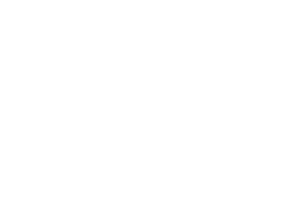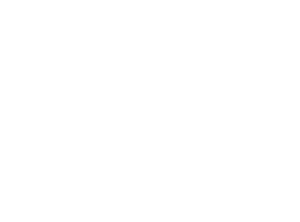
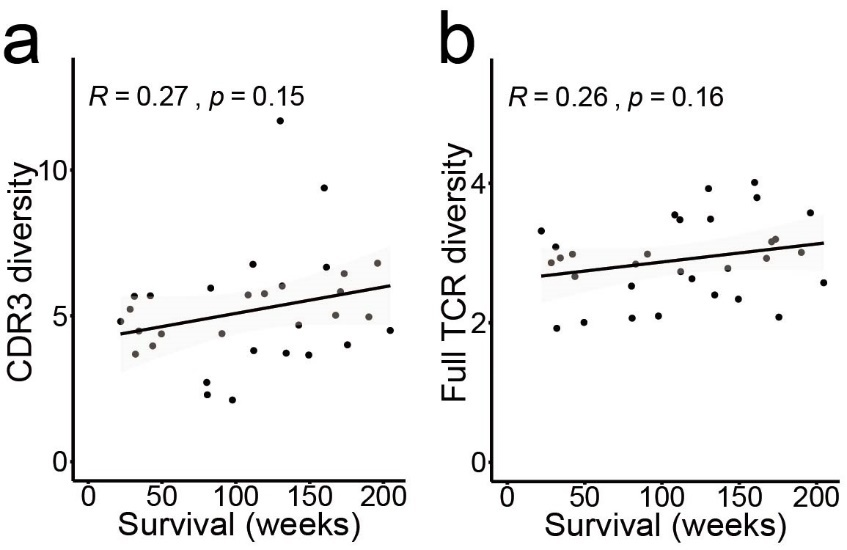
дљЬиАЕй¶ЦеЕИжµЛйЗПдЇЖCDR3еЇПеИЧзЪДе§Ъж†ЈжАІпЉМеєґе∞ЖMHCзЫЄдЇТдљЬзФ®еМЕжЛђеЬ®еЖЕпЉМжµЛйЗПдЇЖеЕ®йХњTCRгАВеЬ®дЄ™дљУж∞іеє≥дЄКпЉМиЩљзДґзЉЇдєПзїЯиЃ°е≠¶жДПдєЙпЉМдљЖиІВеѓЯеИ∞TCRе§Ъж†ЈжАІдЄОжВ£иАЕжАїдљУзФЯе≠ШзОЗдєЛйЧіе≠ШеЬ®еЉ±ж≠£зЫЄеЕ≥(и°•еЕЕеЫЊ2)гАВ
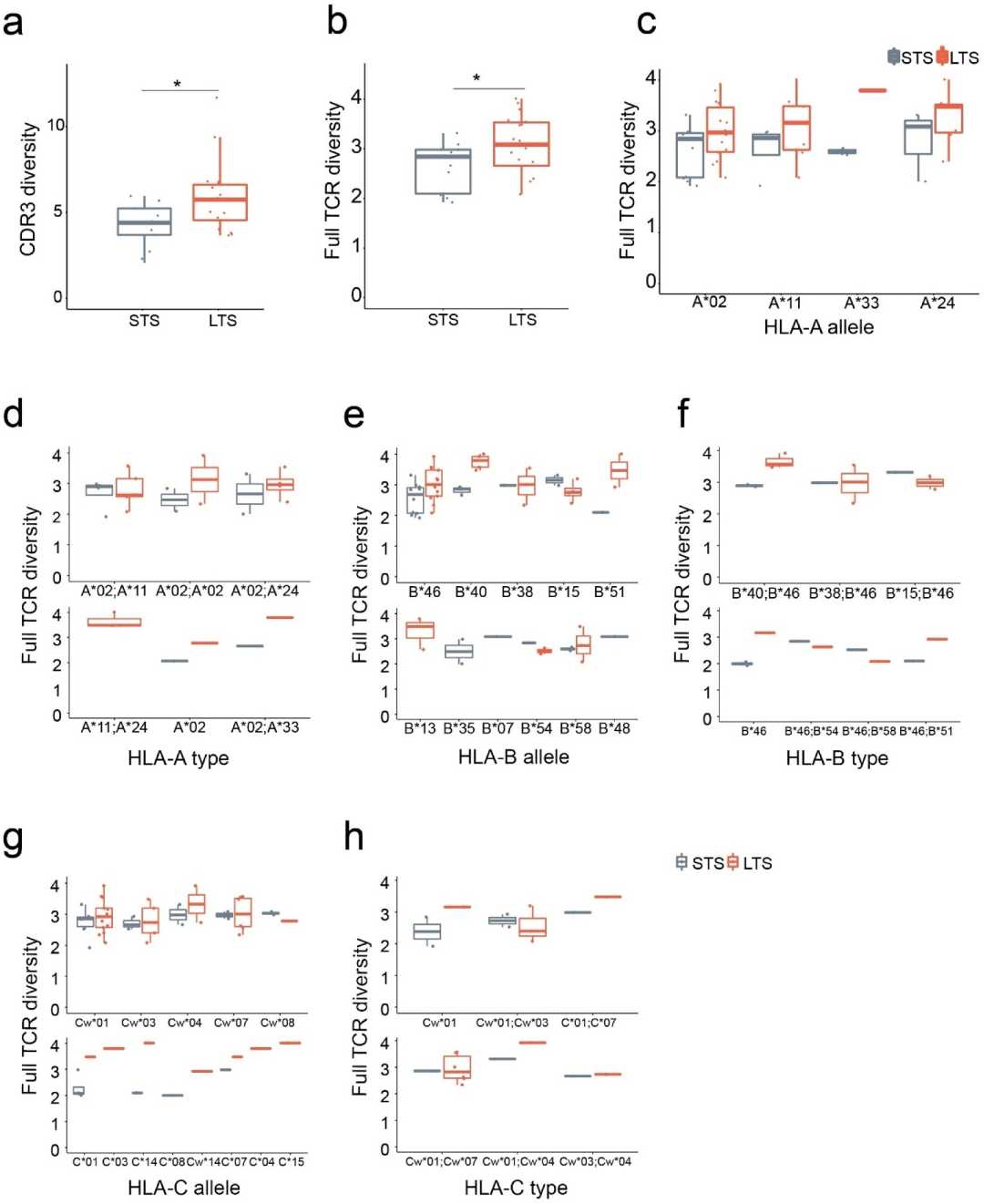
еЫЊ1.EBVзЙєеЉВжАІињЗзїІTзїЖиГЮж≤їзЦЧзЪДйХњжЬЯеєЄе≠ШиАЕеЕЈжЬЙжЫійЂШзЪДTCRеЇУе§Ъж†ЈжАІпЉМж≤°жЬЙHLAеБПиІБ
ж≥®пЉЪa.CDR3еМЇеЯЯLTSдЄОSTSзЪДShannonзЖµжѓФиЊГгАВ
b.еЕ®йХњTCRзЪДShannonзЖµжѓФиЊГгАВ
c-h.е≠РзїДдЄ≠зЪДй¶ЩеЖЬзЖµжѓФиЊГгАВLTSеТМSTSж†єжНЃеЕґHLAз≠ЙдљНеЯЇеЫ†жИЦз±їеЮЛињЫи°МеИЖзїДгАВжѓПдЄ™зВєдї£и°®жѓПдљНжВ£иАЕCTLеЇУзЪДй¶ЩеЖЬзЖµеАЉгАВPеАЉйЗЗзФ®йЭЮеПВжХ∞Wilcoxonзђ¶еПЈзІ©ж£Ай™МиЃ°зЃЧ;*пЉМp < 0.05гАВLTSпЉМйХњжЬЯеєЄе≠ШиАЕ;STSпЉМзЯ≠жЬЯеєЄе≠ШиАЕгАВ
зДґиАМпЉМеЬ®е∞ЖжВ£иАЕеИЖдЄЇSTSзїДеТМLTSзїДеРОпЉМеЬ®CDR3еТМеЕ®йХњжµЛйЗПдЄ≠пЉМиЊУж≥®еИ∞LTSзЪДCTLеЕЈжЬЙжШОжШЊжЫійЂШзЪДTCRе§Ъж†ЈжАІ(еЫЊ1a, 1b)гАВ
е∞љзЃ°ж†ЈжЬђйЗПйЩРеИґдЇЖжЯРдЇЫдЇЪзїДзЪДзїЯиЃ°е≠¶жДПдєЙпЉМдљЖеѓєдЇОеЗ†дєОжЙАжЬЙHLAеЬ®дЄНеРМз±їеЮЛдЄ≠пЉМLTSзїДзЪДTCRеЇУе§Ъж†ЈжАІжЫійЂШ(еЫЊ1c-h)пЉМи°®жШОиІВеѓЯеИ∞зЪДCTLеЇУе§Ъж†ЈжАІзЉЇдєПHLAеБПеАЪгАВжАїдєЛпЉМињЩдЇЫеПСзО∞и°®жШОпЉМеЬ®CTLзФЯдЇІињЗз®ЛдЄ≠пЉМе§Ъж†ЈеМЦEBVзЙєеЉВжАІTзїЖиГЮеЕЛйЪЖзЪДдЇІзФЯдЇЖеПѓиГљжЬЙеИ©дЇОжКЧNPCзЪДеКЯжХИгАВ
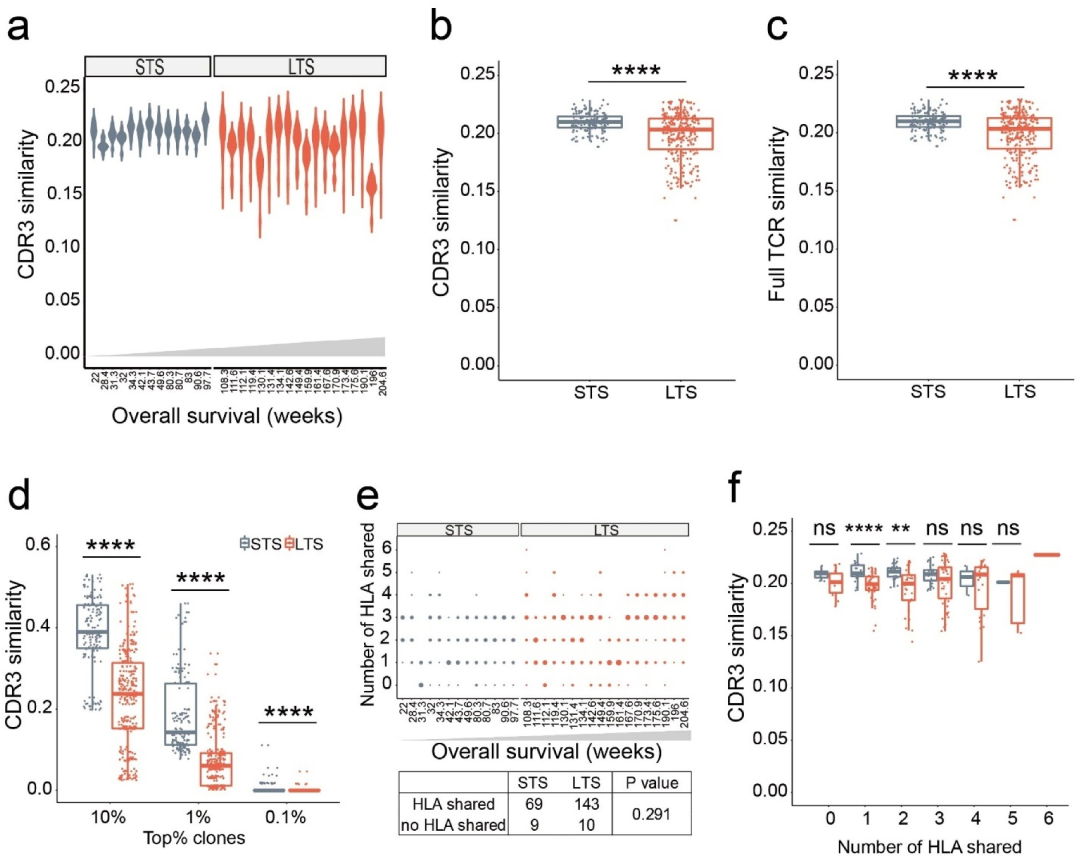
е∞љзЃ°е§Ъж†ЈжАІеИЖжЮРи°®жШОпЉМеПѓиГљдЄНе≠ШеЬ®жФѓйЕНCLTжКЧиВњзШ§еПНеЇФзЪДжШЊжАІEBVжКЧеОЯпЉМдљЖе∞ЪдЄНжЄЕж•ЪеЬ®LTSдЄ≠CLTжШѓеР¶йТИеѓєдЄАзїДеЕ±еРМзЪДвАЬжЬЙжХИвАЭжКЧеОЯжПРеЗЇгАВдљњзФ®JaccardжМЗжХ∞еИЖжЮРпЉМдљЬиАЕйЗПеМЦдЇЖзїДеЖЕеТМжВ£иАЕйЧіTCRеЇУзЪДзЫЄдЉЉжАІгАВдЄОSTSзїДзЫЄжѓФпЉМLTSзїДжВ£иАЕйЧіCTL CDR3зЫЄдЉЉжАІиЊГдљО(еЫЊ2a, 2b)пЉМеєґдЄФпЉМељУиѓ•еИЖжЮРжЙ©е±ХеИ∞жХідЄ™TCRжЧґпЉМиІВеѓЯеИ∞зЫЄеРМзЪДж®°еЉП(еЫЊ2c)гАВ
дљЬиАЕињШиІВеѓЯеИ∞пЉМжѓПдЄ™CTLдЇІзЙ©зЪДеЕЛйЪЖйҐСзОЗеИЖеЄГеЬ®еЊИе§Із®ЛеЇ¶дЄКеБПеРСдЇОйЂШеЇ¶жЙ©еҐЮзЪДеЕЛйЪЖ:пЉЪйҐСзОЗжОТеРНеЬ®еЙН1%зЪДеЕЛйЪЖеЮЛзіѓиЃ°еН†LTSеЇУзЪД90.10%(¬±12.85%)пЉМSTSеЇУзЪД96.83%(¬±1.41%)гАВеѓєдЇОж†ЈжЬђйЧіеЕЛйЪЖеЮЛзЫЄдЉЉжАІжµЛйЗПпЉМињЩзІНеИЖеЄГеЙЦйЭҐеПѓиГљдљОдЉ∞дЇЖйЂШйҐСеЕЛйЪЖзЪДиі°зМЃгАВдЄЇдЇЖеЗПиљїињЩзІНеБПиІБпЉМдљЬиАЕиѓДдЉ∞дЇЖжЬАжЩЃйБНзЪДеЕЛйЪЖеЮЛдєЛйЧізЪДзЫЄдЉЉжАІгАВйАЪињЗеѓєеЙН10%гАБеЙН1%еТМеЙН0.1%еМЇйЧізЪДжЬАйЂШйҐСзОЗеЕЛйЪЖзЪДи∞ГжЯ•пЉМеПСзО∞LTS(еє≥еЭЗзЫЄдЉЉеАЉдЄЇ0.2292 ~ 0.0006)еТМSTS(еє≥еЭЗзЫЄдЉЉеАЉдЄЇ0.3868 ~ 0.0051)жВ£иАЕйЧіTCR CDR3зЫЄдЉЉжАІйАРжЄРйЩНдљОпЉМи°®жШОиЊГйЂШйҐСзОЗеЕЛйЪЖеѓєзЫЄдЉЉжАІзЪДиі°зМЃжЫіе§ІгАВзДґиАМпЉМдЄОSTSзЫЄжѓФпЉМеЬ®жѓПдЄ™жОТеРНз±їеИЂдЄ≠пЉМLTSзїДзЪДCDR3еЕ±дЇЂзОЗжШОжШЊиЊГдљО(еЫЊ2d)гАВ
жАїдєЛпЉМињЩдЇЫзїУжЮЬи°®жШОпЉМеЬ®LTSдЄ≠пЉМиЊУж≥®зЪДCTLзїЖиГЮдЄН姙еПѓиГљйТИеѓєдЄАзїДеЕ±еРМзЪДи°®дљНжЙ©еҐЮгАВдЄОдЄКињ∞жВ£иАЕеЖЕйГ®TCRе§Ъж†ЈжАІеИЖжЮРдЄАж†ЈпЉМињЩдЇЫжВ£иАЕйЧіCDR3зЫЄдЉЉжАІеИЖжЮРдєЯи°®жШОпЉМеЕ±еРМзЪДEBVжКЧеОЯеПѓиГљдЄНжШѓжКЧNPC CTLдЄіеЇКзЦЧжХИзЪДдЄїи¶БеЖ≥еЃЪеЫ†зі†гАВ
дЄЇдЇЖз°ЃеЃЪињЩзІНзЫЄдЉЉжАІеИЖжЮРжШѓеР¶еПЧеИ∞зїДеЖЕHLAйЗНеП†зЪДељ±еУНпЉМдљЬиАЕж£АжЯ•дЇЖеЇУзЫЄдЉЉжАІеТМHLAеЕ±дЇЂдєЛйЧізЪДзЫЄеЕ≥жАІпЉМдЄЙдЄ™з≠ЙдљНеЯЇеЫ†ж£АжµЛIеЮЛHLAпЉМдЄ™дљУйЧіеЕ±жЬЙHLAз≠ЙдљНеЯЇеЫ†жХ∞дЄЇ0 ~ 6дЄ™гАВLTSзїДеТМSTSзїДHLAе§НжЭВжАІжЧ†еЈЃеЉВ(еЫЊ2e)гАВж≠§е§ЦпЉМжЧ†иЃЇHLAе§Ъж†ЈжАІе¶ВдљХпЉМжЙАжЬЙSTSзЪДCDR3еЕ±дЇЂзОЗйГљйЂШдЇОLTS(еЫЊ2f)гАВ
дЄЇдЇЖињЫи°МMotifеИЖжЮРпЉМдљЬиАЕе∞ЖжЙАжЬЙLTSдЄ≠зЪДжЙАжЬЙCDR3еЕЛйЪЖеЮЛж±ЗйЫЖеЬ®дЄАиµЈпЉМеєґйАЪињЗеОїйЩ§йЗНе§НеЇПеИЧзФЯжИРеФѓдЄАзЪДCDR3еИЧи°®гАВCDR3еЕЛйЪЖеЮЛеЙН10%зЪД2merгАБ3merеТМ4merињЮжО•ж∞®еЯЇйЕЄеЇПеИЧжЮДжИРдЇЖеИЭеІЛеЯЇеЇПеЇУгАВиЃ°зЃЧLTSзїДеТМSTSзїДйЧіеРЂжЬЙK-merеЯЇеЇПзЪДCDR3зЪДзїДеЖЕйҐСзОЗдєЛеТМдєЛжѓФ(LTS/STS)гАВзДґеРОпЉМдљЬиАЕеИ©зФ®еЗЖз°ЃжАІзЃЧж≥ХжЭ•иѓЖеИЂеЬ®LTSзЪДCLTдЄ≠дЉШеЕИеѓМйЫЖзЪДеЯЇеЇПпЉМеЕґдЄ≠еЗЖз°ЃжАІеЃЪдєЙдЇЖиЊУеЕ•еПШйЗПйҐДжµЛзїУжЮЬзЪДз®ЛеЇ¶(еЫЊ3a)гАВ
ељУLTS/STSзЪДжѓФеАЉе§ІдЇО10жЧґпЉМеЕ±жЬЙ11дЄ™еЯЇеЇП襀йЙіеЃЪдЄЇдЄОжАїзФЯе≠ШзОЗйЭЮиіЯзЫЄеЕ≥(еЫЊ3a, 3b)гАВ
дї•ињЩ11дЄ™еЯЇеЇПзЪДе≠ШеЬ®еТМйҐСзОЗдљЬдЄЇеФѓдЄАзЪДеИЖзїДеПВжХ∞пЉМдљЬиАЕе∞Же§Іе§ЪжХ∞жВ£иАЕеИЖеЉАдї•еМєйЕНдїЦдїђзЪДдЄіеЇКеПНеЇФпЉМеП™жЬЙ3дЄ™LTS襀йФЩиѓѓиѓЖеИЂ(еЗЖз°ЃзОЗдЄЇ90.3%;еЫЊ3c)пЉМи°®жШОйАЪињЗжЬЇеЩ®е≠¶дє†иѓЖеИЂзЪДCDR3еЯЇеЇПеПѓдї•жЬЙжХИеЬ∞еѓєеПЧзЫКдЇОEBV-CTLж≤їзЦЧзЪДNPCжВ£иАЕињЫи°МеИЖе±ВгАВињСеєіжЭ•пЉМиЃЄе§ЪEBVзЙєеЉВжАІTCR襀еКЯиГљй™МиѓБеєґе≠ШеЕ•еЕђеЕ±жХ∞жНЃеЇУпЉМе¶ВVDJdbеТМMcPAS-TCRгАВ
дљЬиАЕе∞ЖжЙАжЬЙињЩ11дЄ™дЄОеПНеЇФзЫЄеЕ≥зЪДдЄїйҐШжПРдЇ§еИ∞ињЩдЄ§дЄ™жХ∞жНЃеЇУдЄ≠ињЫи°МжРЬ糥гАВеЬ®жКЧEBVжКЧеОЯи°®дљНзЪДTCR(s) CDR3еМЇжИРеКЯеПСзО∞дЇЖDGAGгАБEESгАБEVAGгАБGSRSгАБKTGEгАБLYLгАБSPFSеТМTTNT 8дЄ™еЯЇеЇПгАВињЩдЇЫи°®дљНеИЖеЄГеЬ®BMLF1гАБBRLF1гАБEBNAеТМBZLF1иЫЛзЩљдЄК(еЫЊ3d)гАВињЩжФѓжМБдЇЖдЄАзІНиІВзВєпЉМеН≥ињЩдЇЫеЯЇеЇПе±ЮдЇОеЬ®зФЯдЇІињЗз®ЛдЄ≠襀EBVжКЧеОЯжЙ©еҐЮзЪДCTLsгАВ
жО•дЄЛжЭ•пЉМдљЬиАЕеИ©зФ®жЬЇеЩ®е≠¶дє†ињЫдЄАж≠•еѓєињЩ11дЄ™CDR3еЯЇеЇПзЪДйЗНи¶БжАІињЫи°МжОТеЇПгАВдЄЇжѓПдЄ™motifзФЯжИРдЄ§дЄ™зЛђзЂЛзЪДз≤ЊеЇ¶еПВжХ∞пЉЪmean reduction accuracyеТМmean reduction GiniгАВеЙНиАЕеБЗиЃЊеЕ≥йФЃеПШйЗПзЪДжОТеИЧдЉЪйЩНдљОж®°еЮЛз≤ЊеЇ¶пЉМйАЪињЗеѓєжѓПдЄ™еПШйЗПињЫи°МжОТеИЧжЭ•иЃ°зЃЧз≤ЊеЇ¶еПШеМЦгАВеРОиАЕжЮДеїЇеНХдЄ™еЖ≥з≠Цж†СпЉМзДґеРОжµЛйЗПжѓПдЄ™еПШйЗПзЪДиКВзВєжЭВиі®зЪДеє≥еЭЗжАїеЗПе∞СгАВдЄ§зІНжЦєж≥ХйГљз°ЃеЃЪдЇЖзЫЄеРМзЪДдЄ§дЄ™4merеЯЇеЇПпЉМSPFSеТМSPDQпЉМеЃГдїђеѓєжЙАжЬЙжВ£иАЕзЪДеПНеЇФйҐДжµЛеЗЖз°ЃжАІиі°зМЃжЬАе§І(еЫЊ3e)пЉМињЩдЄ§дЄ™еЯЇеЇПеЬ®LTS CTLзЪДTCRдЄ≠йГљжШЊиСЧеѓМйЫЖ(еЫЊ3f)гАВеѓєеЕґдїЦ9дЄ™еЯЇеЇПзЪДйҐСзОЗжѓФиЊГдєЯеПСзО∞LTSзїДзЪДеЯЇеЇПSERRдЄ∞еѓМ(еЫЊS3a)пЉМе∞љзЃ°еЕґеѓєйҐДжµЛз≤ЊеЇ¶зЪДељ±еУНиЊГдљО(еЫЊ3e)гАВдљЬдЄЇзФЯзЙ©ж†ЗењЧзЙ©иѓДдїЈпЉМROCеИЖжЮРеЊЧеЗЇSPFSеТМSPDQзЪДжЫ≤зЇњдЄЛйЭҐзІѓ(AUC)еИЖеИЂдЄЇ89.1%еТМ84%(еЫЊ3g)гАВ
еѓєдЇОдЄ™дљУжВ£иАЕиАМи®АпЉМSPQDеЯЇеЇПзЪДзіѓиЃ°CDR3йҐСзОЗдЄОLTSжВ£иАЕзЪДжАїзФЯе≠ШзОЗеСИж≠£зЫЄеЕ≥(еЫЊ3hеТМеЫЊS3b)гАВж≠§е§ЦпЉМињЩдЄ§дЄ™еЯЇеЇПеЬ®HLAз≠ЙдљНеЯЇеЫ†дЄЇA2гАБA11жИЦB46зЪДLTSдЄ≠жШЊиСЧеѓМйЫЖ(еЫЊ3i)гАВжАїдєЛпЉМињЩдЇЫзїУжЮЬи°®жШОпЉМеЬ®иЊУж≥®зЪДCTLзЪДCDR3еМЇеЯЯеЖЕзЙєеЉВжАІеЯЇеЇПзЪДеѓМйЫЖеПѓиГљйҐДжµЛйЉїеТљзЩМжВ£иАЕеѓєEBV-CTLж≤їзЦЧзЪДеПНеЇФгАВеЕЈдљУжЭ•иѓіпЉМйАЪињЗжХ∞жНЃеЇУжРЬ糥пЉМз°ЃеЃЪSPFSеЯЇеЇПе±ЮдЇОиѓЖеИЂBLZF1иЫЛзЩљдЄ≠RAKFKQLLи°®дљНзЪДTCRs(еЫЊ3d)гАВеЃЮй™МдЄ≠пЉМHLA-B*08:01еСИйАТдЇЖиѓ•и°®дљНгАВиЩљзДґеЬ®еЯЇеЇПеИЖжЮРдЄ≠ж≤°жЬЙеПСзО∞дїїдљХжВ£иАЕзЪДHLA- b *08:01пЉМдљЖжИСдїђдљњзФ®NetMHCpan4.141еТМHLAthena42зЃЧж≥ХиѓДдЉ∞дЇЖеЕґдїЦHLAз≠ЙдљНеЯЇеЫ†жШѓеР¶еПѓиГље≠ШеЬ®иѓ•и°®дљНгАВињЩдЇЫзФЯзЙ©дњ°жБѓе≠¶еЈ•еЕЈйҐДжµЛRAKFKQLLи°®дљНеПѓиГље≠ШеЬ®HLA-A*33:03гАБB*15:01гАБB*15:25гАБB*38:02жИЦB*46:01пЉМеЕЈжЬЙиґ≥е§ЯзЪДдЇ≤еТМеКЫгАВеЬ®жИСдїђзЪДйШЯеИЧдЄ≠пЉМ31дЊЛжВ£иАЕдЄ≠жЬЙ27дЊЛиЗ≥е∞СеРЂжЬЙдЄАзІНHLAз≠ЙдљНеЯЇеЫ†(и°®S4)гАВеЫ†ж≠§пЉМеѓєдЇОдљЬиАЕзЪДйШЯеИЧпЉМRAKFKQLLи°®дљНеЬ®иВњзШ§зїДзїЗдЄ≠зЪДеСИзО∞жШѓеПѓиГљзЪДгАВињЩжДПеС≥зЭАCTLдЇІзЙ©дЄ≠еРЂжЬЙSPFSзЪДTCRsеПѓиГљжПРдЊЫдЄіеЇКиІВеѓЯеИ∞зЪДжЬЙзЫКжИРеИЖгАВ
пЉЛ
жАїзїУ
EBVйШ≥жАІйЉїеТљзЩМзїЖиГЮи°®иЊЊEBNA1гАБLMP1гАБLMP2з≠ЙеЗ†зІНйАЪзФ®зЧЕжѓТжКЧеОЯпЉМеПѓжЬЙжХИйЭґеРСжЄЕйЩ§иВњзШ§зїЖиГЮгАВе§Ъй°єдЄіеЇКз†Фз©ґи°®жШОпЉМеРЂжЬЙLMP2зЙєеЉВжАІеПНеЇФжАІзЪДEBVеѓМйЫЖCTLдЄОеЃҐиІВеПНеЇФзЫЄеЕ≥гАВе∞љзЃ°зФЯе≠ШеИЖжЮРжШЊз§ЇжАїзФЯе≠ШзОЗжШЊиСЧжПРйЂШпЉМдљЖйАЪињЗELISPOTеИЖжЮРпЉМиЊУж≥®CTLsдЄ≠LMP2зЙєеЉВжАІTзїЖиГЮзЪДйҐСзОЗжЧ†ж≥ХйҐДжµЛдЄ™дљУзЪДжКЧиВњзШ§еПНеЇФгАВеЬ®ињЩйЗМпЉМйАЪињЗжЈ±еЇ¶жµЛеЇПињЫи°МдЇЖйЂШйАЪйЗПTCRќ≤еЇУеИЖжЮРпЉМдї•жЈ±еЕ•дЇЖиІ£EBV-CTLеЇУзЪДзЙєеЊБгАВеПСзО∞LTSеЕЈжЬЙиЊГйЂШзЪДжВ£иАЕеЖЕйГ®TCRеЇУе§Ъж†ЈжАІеТМиЊГдљОзЪДжВ£иАЕйЧізЫЄдЉЉжАІгАВдљњзФ®жЦ∞еЉАеПСзЪДMotif AnalysisзЃЧж≥ХпЉМз°ЃеЃЪдЇЖ11дЄ™иГље§ЯеМЇеИЖLTSеТМSTSзЪДMotifпЉМињЩдЇЫMotifеПѓиГљжЬАзїИзФ®дљЬдЄіеЇКзЦЧжХИзЪДйҐДжµЛеЫ†е≠РгАВ
дљЬиАЕзЪДз†Фз©ґжПРеЗЇдЇЖдЄАзІНжЦ∞зЪДжЦєж≥ХпЉМйАЪињЗдЄОNPCжВ£иАЕзФЯе≠ШзЫЄеЕ≥зЪДCDR3иѓЖеИЂеЯЇеЇПеѓєдљУе§ЦзФЯжИРзЪДEBV-CTLsињЫи°МеИЖе±ВпЉМдї•дЉШеМЦж≤їзЦЧеїЇиЃЃгАВ
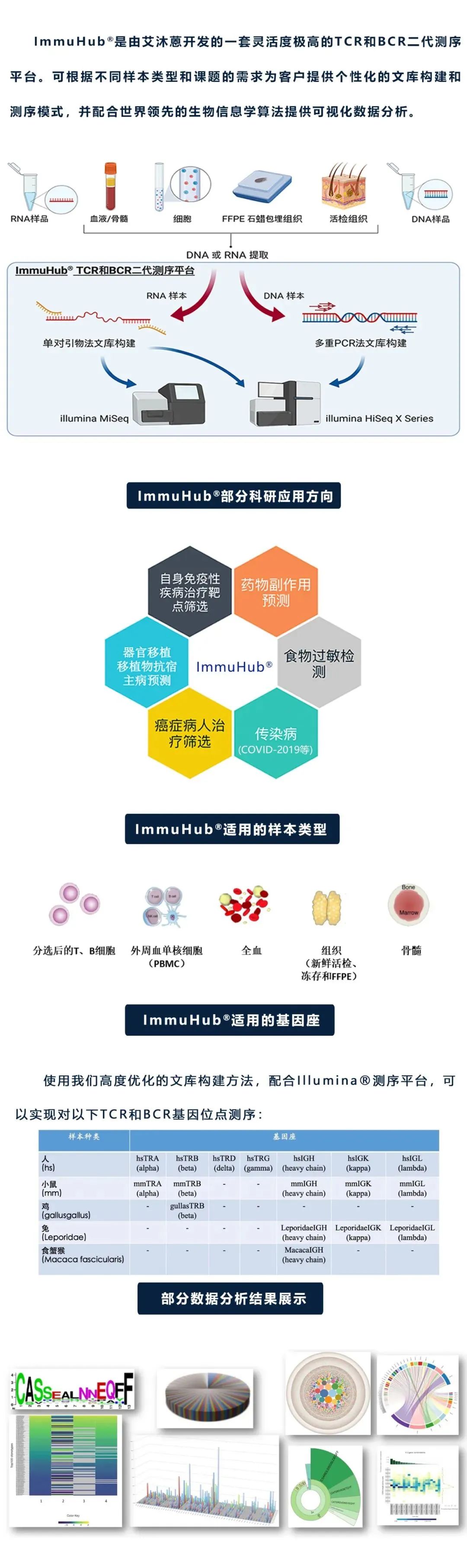

жЭ≠еЈЮиЙЊж≤РиТљзФЯзЙ©зІСжКАжЬЙйЩРеЕђеПЄжИРзЂЛдЇО2016еєіпЉМжШѓеЫљеЖЕеЙНж≤њзЪДдЄУж≥®дЇОеЕНзЦЂеЯЇеЫ†зїДе≠¶жКАжЬѓзЪДеЫљеЃґйЂШжЦ∞жКАжЬѓдЉБдЄЪгАВеИЫеІЛдЇЇеЫҐйШЯжЭ•иЗ™зЊОеЫљиКЭеК†еУ•е§Іе≠¶пЉМеЬ®2010еєіеЉАеІЛдљњзФ®еЕНзЦЂзїДйЂШйАЪйЗПжµЛеЇПжКАжЬѓеЉАе±ХеРДзІНзЦЊзЧЕзЫЄеЕ≥з†Фз©ґпЉМдЇО2016еєійАЪињЗиЗ™дЄїз†ФеПСпЉМеЕ®еЫљй¶ЦеЃґжО®еЗЇNGS-MRDи°Ажґ≤иВњзШ§еЊЃе∞ПжЃЛзХЩзЧЕпЉИMRDпЉЙж£АжµЛSeq-MRD¬ЃпЉМеєґжОИжЭГж≥ЫзФЯе≠РпЉИзЇ≥жЦѓиЊЊеЕЛдї£з†БпЉЪGTHпЉЙдљњзФ®гАВеРМжЧґпЉМеЕђеПЄжЛ•жЬЙImmun-Traq¬ЃиВњзШ§ж≤їзЦЧдЉійЪПиѓКжЦ≠гАБImmun-Cheq¬Ѓ |TзїЖиГЮеЕНзЦЂжµЛиѓДдї•еПКImmuHub¬ЃеЕНзЦЂзїДжµЛеЇПзІСз†ФжЬНеК°дЇІеУБпЉМеєґеЄГе±АжЬЙеЯЇдЇОAIжЬЇеЩ®е≠¶дє†зЃЧж≥ХзЪДT-classifier¬ЃзЦЊзЧЕжЧ©з≠ЫгАБеНХзїЖиГЮжµЛеЇПгАБTCR-TиНѓзЙ©еЉАеПСз≠Йеє≥еП∞зЃ°зЇњгАВеЕђеПЄжЮДеїЇеЗ†еНБй°єеПСжШОдЄУеИ©еТМиљѓдїґиСЧдљЬжЭГдЄЇж†ЄењГзЪДиЗ™дЄїзЯ•иѓЖдЇІжЭГдљУз≥їпЉМдЄЇеМїйЩҐдЄіеЇКгАБзФЯеСљзІСе≠¶з†Фз©ґгАБжЦ∞иНѓеЉАеПСз≠ЙжПРдЊЫиІ£еЖ≥жЦєж°ИеТМдЇІеУБгАВ
иЙЊж≤РиТљдЄУж≥®дЇОйАЪињЗиІ£з†БйАВеЇФжАІеЕНзЦЂз≥їзїЯжЭ•жФєеПШзЦЊзЧЕзЪДиѓКжЦ≠еТМж≤їзЦЧпЉМеєґиЗіеКЫдЇОжО®ињЫеЕНзЦЂй©±еК®еМїе≠¶йҐЖеЯЯеПСе±ХгАВ

